Die Proteinstabilität ist entscheidend für den Erfolg oder Misserfolg der Entwicklung eines Biopharmazeutikums. Die Proteinstabilität ist ein wichtiger Parameter hinsichtlich Produktion, Herstellung, Formulierung, Langzeit-Lagerfähigkeit, Verabreichung an den Patienten und Wirksamkeit. Hochstabile Proteine neigen weniger zu Problemen während des Herstellungsprozesses, sind kostengünstiger in der Produktion und bleiben mit größerer Wahrscheinlichkeit während Formulierung und Lagerung funktionsfähig, ohne chemische Veränderung oder Aggregation. Beim QbD-Ansatz (Quality by Design) für die biopharmazeutische Entwicklung ist die Stabilitätscharakterisierung Teil der Bewertung der Entwicklungsfähigkeit und Wirkstofffähigkeit eines potenziellen Wirkstoffkandidaten. Sie wird auch während der Verfahrensentwicklung und Herstellung durchgeführt. Die Stabilitätsdaten fließen zudem in die Charakterisierung der höheren Ordnungsstruktur (HOS) und das Fingerprinting ein. Diese Daten werden zur Unterstützung der Herstellung sowie für Biokomparabilitäts- und Biosimilaritätsstudien herangezogen. Auch bei den Zulassungsanträgen für neue biopharmazeutische Wirkstoffe und Biosimilars wird die HOS-Charakterisierung von Proteinen zunehmend vorausgesetzt.
Aufgrund des komplexen Charakters von Proteinen spielen biophysikalische Werkzeuge bei der vollständigen Charakterisierung eines biopharmazeutischen Produkts eine wichtige Rolle. Zur Bewertung der Proteinstabilität stehen verschiedene biophysikalische Werkzeuge zur Verfügung. Hierzu gehören u. a. die CD-Spektroskopie (CD), die dynamische und statische Lichtstreuung (DLS und SLS), die Größenausschlusschromatographie mit Mehrwinkel-Lichtstreudetektoren (SEC-MALS), die Fourier-Transformations-Infrarotspektrometrie (FTIR), die analytische Ultrazentrifugation (AUC), die Größenausschlusschromatographie (SEC), die Differential-Scanning-Fluorimetrie (DSF), die intrinsische Fluoreszenz (IF) und die dynamische Differenzkalorimetrie (DSC).
Wenngleich all diese biophysikalischen Assays in der biopharmazeutischen Entwicklung eine wichtige Rolle spielen, kommt der Charakterisierung der thermischen Stabilität mittels DSC eine besondere Bedeutung zu. Ein Artikel aus dem Jahr 2015 über biophysikalische Methoden zur Charakterisierung der höheren Ordnungsstruktur monoklonaler Antikörper (Gokarn et al.) stellt fest: „Die DSC ist nach wie vor ein konkurrenzloses Verfahren zur Beurteilung der thermodynamischen Stabilität von Proteinen unter gegebenen Pufferbedingungen.“[1]
Das vorliegende Whitepaper konzentriert sich auf den Einsatz der DSC zur Charakterisierung der thermischen Stabilität von Protein-Biopharmazeutika (v. a. von Antikörpern) sowie als HOS-Charakterisierungswerkzeug für biopharmazeutische Vergleichbarkeitsstudien (Chargenvergleich, Auswirkung von Prozessänderungen usw.) und für die Entwicklung von Biosimilars.
Die DSC ist ein mikrokalorimetrisches Verfahren, mit dem die thermische und konformationelle Stabilität von Proteinen, Nukleinsäuren, Lipiden und anderen Biopolymeren charakterisiert werden kann.[2-7] Die DSC misst die Wärmekapazität als Funktion der Temperatur. Bei den für die Proteincharakterisierung eingesetzten DSC-Geräten, wie sie in diesem Whitepaper beschrieben werden, handelt es sich um Leistungsdifferenzkalorimeter. Sie verfügen über eine ortsfeste Probenzelle, die das Biopolymer in Lösung enthält, und eine gleichartige Referenzzelle, die mit Pufferlösung befüllt ist. Der für die Probenzelle gemessene Wärmekapazitätswert (Cp) wird mit dem der Referenzzelle verglichen. Während die Temperatur der Zellen erhöht wird, wird der Temperaturunterschied zwischen Referenz- und Probenzelle kontinuierlich gemessen und in Leistungswerte umgewandelt. Die DSC ist ein sogenannter Forced-Degradation-Assay: Das Protein wird einer steigenden Temperatur ausgesetzt, wodurch es beginnt, sich zu entfalten. Dabei nimmt der Cp-Wert des Proteins zu (Abbildung 1).
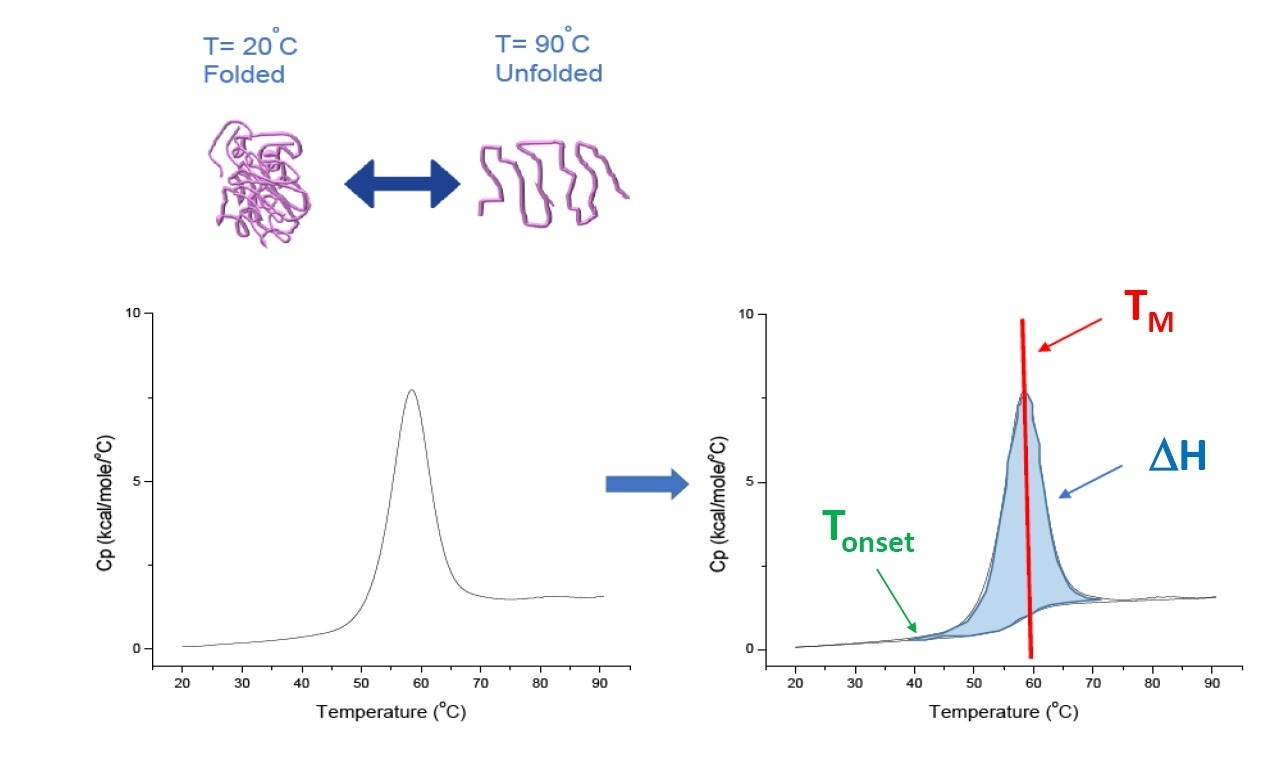
Abbildung 1: Funktionsweise der DSC. Die Wärmekapazität (Cp) ändert sich mit der thermischen Denaturierung des Proteins. Der DSC-Versuch beginnt bei einer Temperatur, bei der das Protein vorwiegend in seiner nativen Konformation gefaltet ist. Bei steigender Temperatur beginnt das Protein ab einem bestimmten Punkt, sich zu entfalten bzw. zu denaturieren (Tonset), und der Cp-Wert nimmt zu. Bei der Temperatur, bei der 50 % des Proteins in der nativen Konformation vorliegen und 50 % denaturiert sind, erreicht der Cp-Wert sein Maximum. Dies ist der Mittelpunkt des thermischen Übergangs, TM. Oberhalb von TM ist das Protein vorwiegend denaturiert, und zum Ende des DSC-Versuchs liegt das Protein vollständig in entfalteter Konformation vor. Zu den Parametern des DSC-Versuchs gehören Tonset, TM und die Entfaltungsenthalpie (ΔH).
Die DSC misst die Änderung der Wärmekapazität direkt, ohne dass eine Fluoreszenzmarkierung oder eine andere Markierung oder Sonde benötigt wird. Bei reversibel denaturierten Proteinen wird der Mittelpunkt des thermischen Übergangs (TM) auch Schmelz- oder Denaturierungstemperatur genannt. Bei dieser Temperatur liegen 50 % des Proteins in nativer (gefalteter) Konformation und 50 % in denaturierter Konformation vor. Der TM-Wert ist im DSC-Thermogramm als Peak zu erkennen.
TM ist ein guter Indikator für die thermische Stabilität – je höher der TM-Wert, desto thermisch stabiler ist das Protein. DSC-Thermogramme von Multidomänen-Proteinen (wie z. B. von Antikörpern) weisen typischerweise mehrere Peaks auf, anhand derer für jede Domäne ein TM-Wert ermittelt werden kann (siehe Abbildung 2).
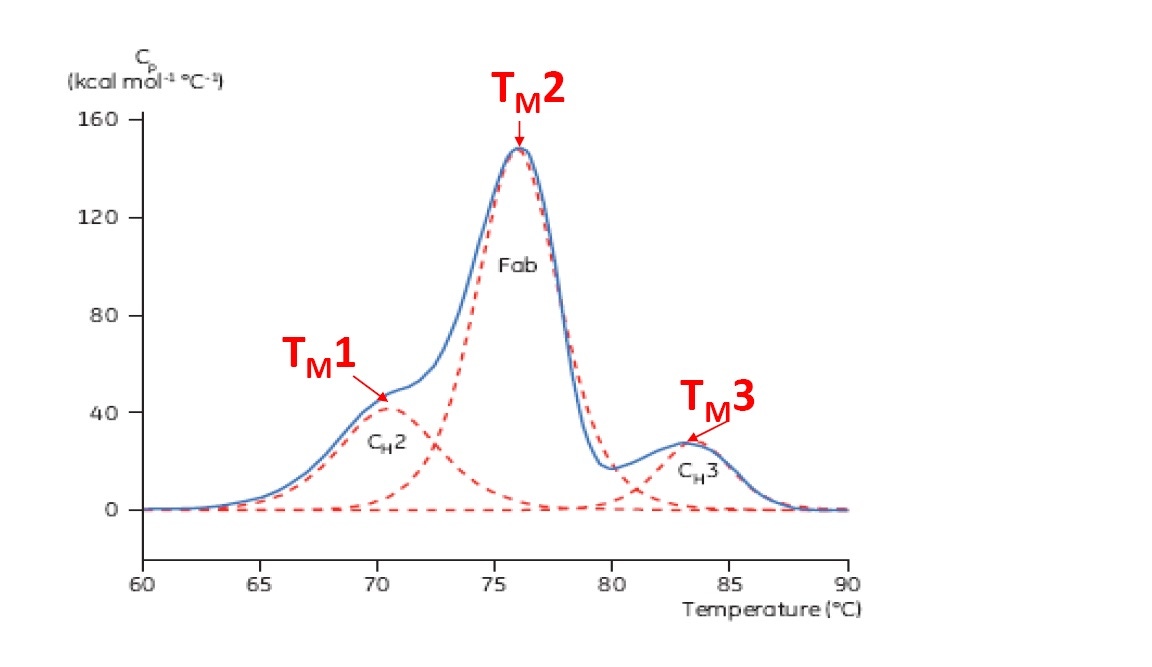
Abbildung 2: Repräsentatives DSC-Thermogramm eines monoklonalen Antikörpers mit Kennzeichnung der CH2-, Fab- und CH3-Domäne. Die gestrichelten roten Linien stellen die dekonvolutierten Peaks der drei einzelnen Domänenübergänge dar. Hierzu sind die jeweiligen TM-Werte gekennzeichnet.
Die DSC liefert auch weitere nützliche Parameter, die zur Charakterisierung und Klassifizierung der Proteinstabilität herangezogen werden können. Hierzu gehört die Entfaltungsenthalpie (ΔH), die proportional zur Fläche unter der Kurve ist. Die Proteinentfaltung verläuft endotherm, da eine Energiezufuhr benötigt wird, um die nicht kovalenten sekundären Bindungen aufzubrechen, die das Protein gefaltet halten. Bestimmt werden können mit der DSC zudem die Werte von Tonset (Beginn der Entfaltung), ΔCp (Wärmekapazitätsänderung bei Entfaltung) und T1/2 (Peakbreite bei halber Peakhöhe, charakteristisch für die Form des Entfaltungsthermogramms). Mittels DSC-Analyse können diese Parameter in beliebiger Kombination bestimmt werden.
Die meisten Proteine denaturieren irreversibel und neigen nach thermischer Denaturierung zur Aggregation oder Präzipitation. Der TM-Wert und weitere, mittels DSC-Analyse irreversibel denaturierter Proteine bestimmte Parameter sind keine echten thermodynamischen Parameter. Die Klassifizierung der TM-Werte aus der DSC-Analyse einer irreversiblen Proteindenaturierung ergibt jedoch einen sehr nützlichen qualitativen Parameter für Stabilitätsuntersuchungen.
Malvern Instruments liefert das System MicroCal VP-Capillary DSC[8,9], ein automatisiertes DSC-Kalorimeter für TM-Untersuchungen und die thermodynamische Charakterisierung von Proteinen und Biopolymeren in Lösung.
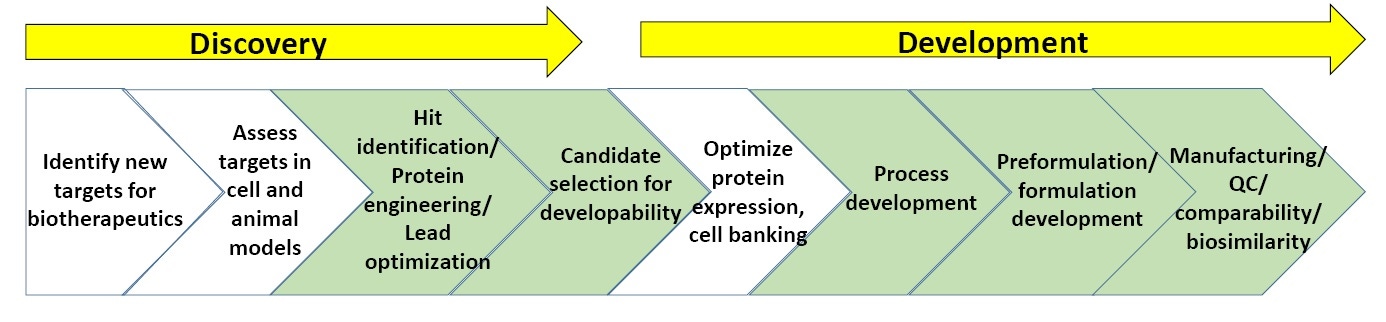
Abbildung 3: Ein allgemeines Schema der Phasen in der Entdeckung und Entwicklung von Biopharmaka
Abbildung 3 zeigt ein allgemeines Schema der biopharmazeutischen Forschungs- und Entwicklungsprozesse. In den grün gekennzeichneten Phasen wird die biophysikalische Charakterisierung (Stabilitätsassays eingeschlossen) am häufigsten eingesetzt. (Am Ende dieses Whitepapers finden Sie eine Liste empfohlener Literatur zur biopharmazeutischen Forschung und Entwicklung).
Bei der Entwicklung eines gewünschten Biopharmazeutikums suchen die Forscher zunächst nach Biomolekülen, die bereits bei der Kandidatenauswahl eine hohe Stabilität zeigen. Eine weitere Erhöhung der Stabilität muss u. U. durch Protein-Engineering erzeugt werden. Während der Aufreinigungsphase wird das Protein aus Bedingungen entfernt, in denen es stabil, korrekt gefaltet und aktiv ist. Es ist daher sehr wichtig, geeignete Puffer, Additive, Aufreinigungsverfahren und Lagerungsbedingungen zu verwenden, um das Protein in dieser Phase so stabil wie möglich zu halten.
Subkutane Formulierungen von Proteinwirkstoffen müssen bei sehr hohen Proteinkonzentrationen (z. T. bei 100 mg/ml) in den Behältnissen (z. B. Injektionsfläschchen oder Fertigspritzen) über Jahre hinweg stabil und unverändert bleiben. Wenn Proteinmoleküle Belastungen ausgesetzt werden (Wärme, Chemikalien, pH-Änderungen, Druck, Vermischung und hoher Konzentration), wie sie in der Formulierung und Produktion von Biopharmaka sämtlich auftreten können, kann die Proteinkonformation das denaturierte (entfaltete) Protein begünstigen. Proteine in Lösung sind zudem empfindlich gegenüber Modifikationen wie Deamidierung und Oxidation, die ebenfalls zu denaturierten, inaktiven Proteinen führen. Bei Protein-Biopharmazeutika können eine Denaturierung oder andere Modifikationen zur Bildung von Aggregaten führen. Diese weisen eine verminderte Wirksamkeit auf oder sind als Wirkstoff funktionslos. Noch kritischer ist, dass die Proteinaggregation potenziell lebensbedrohliche immunogene Reaktionen des Patienten bewirken kann. Die Verwendung stabiler Proteine als Basis für Biopharmazeutika führt zu einer kostengünstigeren Produktion und zu erfolgreichen, wirksamen und sicheren Arzneimitteln.
Die Reihenfolge der Aminosäuren in einer Polypeptidkette, Primärstruktur (1°) genannt, ist die unterste Ebene der Proteinstruktur. Darüber hinaus ist es wichtig, die dreidimensionale Struktur des Proteins zu verstehen und zu charakterisieren. Diese wird auch höhere Ordnungsstruktur (HOS) genannt. Es existieren drei Ebenen der höheren Ordnungsstruktur (HOS) eines Proteins: Die Sekundärstruktur (2°) – sie beschreibt die lokalen Faltungsmuster der Primärstruktur des Proteins, wie die α-Helix, das β-Faltblatt, Schleifen und statistische Knäuel. Die Tertiärstruktur (3°) – sie beschreibt die endgültige 3D-Struktur eines Proteins, die aus einer Anordnung von Sekundärstrukturelementen gebildet wird. Die Quartärstruktur (4°) – sie beschreibt Strukturen, die infolge der Wechselwirkungen zwischen zwei oder mehreren, identischen oder verschiedenen Polypeptidketten entstehen.
Die DSC liefert ein Maß der konformationellen Stabilität und der Änderungen der Tertiär- und Quartärstruktur, die auftreten, wenn ein Protein thermisch denaturiert ist. Zudem liefert sie Hinweise über den Einfluss intrinsischer und extrinsischer Faktoren auf die Proteinstabilität. Die DSC gilt als der qualitativ wie quantitativ beste Assay für die thermische Stabilität. Sie wird zur Charakterisierung von biopharmazeutischen Proteinen sowie zur Vorhersage der Langzeitstabilität eingesetzt.[1,10-14] Die mittels DSC ermittelten TM-Werte werden häufig als Parameter herangezogen, um die Stabilität bei der Kandidatenauswahl (hinsichtlich Entwicklungsfähigkeit), beim Formulierungsscreening und bei der Verfahrensentwicklung zu klassifizieren. Dabei gilt, dass stabilere Proteine einen höheren TM-Wert aufweisen. Auch die mittels DCS gemessenen Größen Enthalpie (ΔH), Tonset, T1/2 und ΔCp werden für die Stabilitätsklassifizierung, die Validierung von DSC-Daten, die quantitative Analyse der Proteinentfaltung und das HOS-Fingerprinting verwendet.[10-14]
DSC-Analysen von Proteinen unter definierten Lösungsbedingungen sind quantitativ und reproduzierbar, wenn die Proteine gleich oder sehr ähnlich sind (Abbildung 4). Das heißt, die DSC-Thermogramme weisen reproduzierbare Verläufe auf und die Parameter (einschließlich TM, ΔH und Tonset) liegen innerhalb eines bestimmten Streubereichs.[12-14] Falls die verglichenen Thermogramme unterschiedlich sind und sich die mittels DSC ermittelten Parameter ändern, weist dies auf ein Ereignis hin, das sich auf die konformationelle Stabilität auswirkt. Dazu gehören Proteinfehlfaltung, Degradation, Aggregation, Lösungsmitteländerungen, posttranslationale Modifikation oder andere Änderungen der höheren Ordnungsstruktur.
Aufgrund der reproduzierbaren und quantitativen Ergebnisse ist die DSC ein wichtiger Assay für Vergleichbarkeits- und HOS-Analysen. Eingesetzt werden kann sie für die Produktevaluierung bei der Herstellung (einschließlich von Chargen- und Standortvergleichbarkeit), den Vergleich von Proteinvarianten und modifizierten Produkten (einschließlich von strukturellen Veränderungen durch Glykosylierung und Oxidation) und Biosimilaritätsstudien. Im Rahmen der HOS-Charakterisierung finden DSC-Daten auch Eingang in Dokumente im regulatorischen Umfeld im Rahmen der Zulassung neuer Wirkstoffe und Biosimilars. Bei einer Befragung von Forschern in der biopharmazeutischen Industrie stuften diese die DSC als „sehr hilfreiches“ bis „äußerst hilfreiches“ biophysikalisches HOS-Werkzeug bei Kandidatenauswahl, Formulierungsentwicklung, Produktcharakterisierung, Verfahrensentwicklung sowie Vergleichbarkeits- und Biosimilaritätsstudien ein.[15]
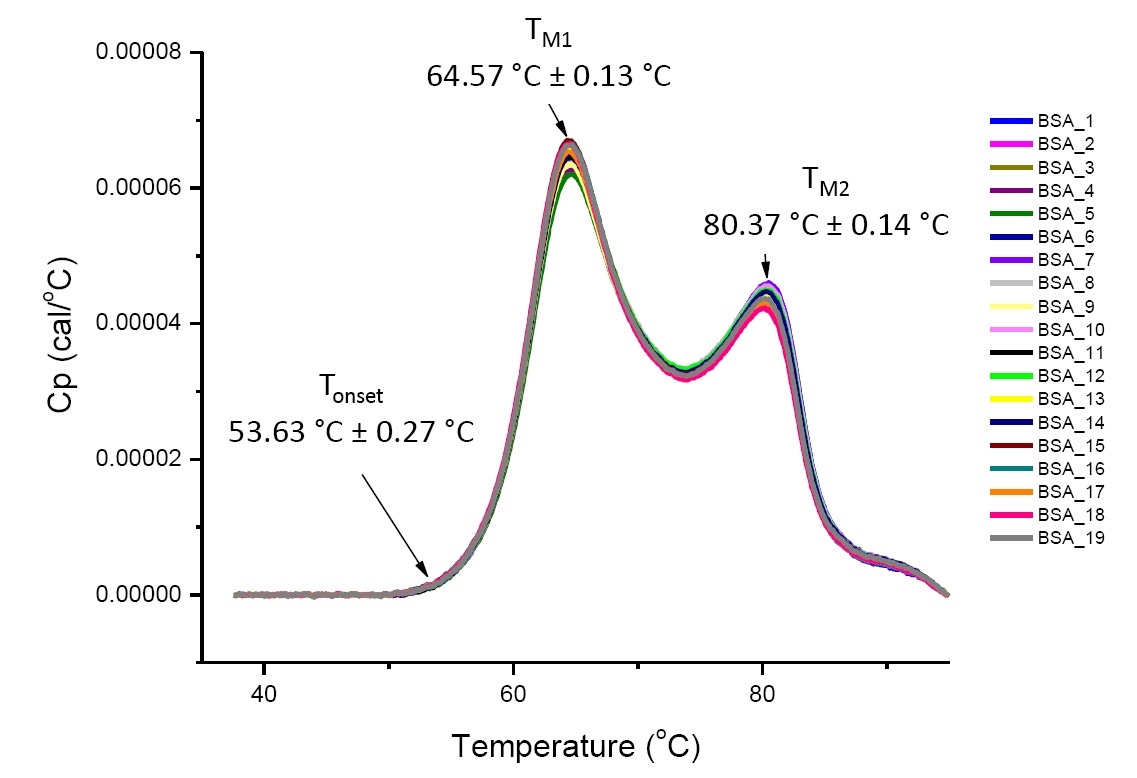
Abbildung 4: Neunzehn DSC-Thermogramme von Rinderserumalbumin (Sigma A1933, chromatographisch gereinigt) in PBS. Auftrag der DSC-Daten nach Normalisierung der Durchlaufrate, Puffer-Puffer-Subtraktion und Subtraktion der Integrationsbasislinie. Angegeben sind Mittelwert und Standardabweichung von Tonset, TM1 und TM2.
Die DSC-Thermogramme von Multidomänenproteinen, wie z. B. von Antikörpern, weisen mehrere Entfaltungsübergänge auf (siehe Abbildung 2). Mit der DSC können die einzelnen Domänen charakterisiert und quantifiziert werden. Ebenso können die individuellen TM-Werte der einzelnen Übergänge bestimmt werden. Die TM-Werte entsprechen den Peaks im Thermogramm. Somit können sie einfach aus den DSC-Daten bestimmt werden, ohne dass eine komplexe Datenanalyse notwendig ist. Alternative biophysikalische Assays zur Bestimmung der TM-Werte, wie z. B. CD, IF und DSF, können bei Multidomänenproteinen nur den ersten TM-Wert detektieren (der bei der niedrigsten Temperatur auftritt) oder den „dominantesten“ TM-Wert. Um aus spektroskopischen Daten oder Fluoreszenzdaten mehr als einen TM-Wert zu extrahieren; werden komplexe Datenanpassungen benötigt. Zudem ist diese Bestimmung u. U. nicht reproduzierbar.
Im Vergleich zu anderen Assays zur TM-Bestimmung wird für die DSC mehr Proteinprobenmaterial pro Durchlauf benötigt. Zudem kann der Durchsatz geringer ausfallen. Bei begrenzter Probenmaterialmenge kann daher zunächst eine TM-Klassifizierung mittels DSF oder IF durchgeführt werden. Anschließend können mehrere Proben ausgewählt werden, um die TM-Werte mittels DSC zu validieren. Bei der Stabilitätsbewertung ist es wichtig, die TM-Ergebnisse mittels DSC zu validieren und sich bei der TM-Berechnung nicht allein auf Fluoreszenz- oder Spektroskopieverfahren zu verlassen. Bei fluoreszenzbasierten Assays treten häufig Artefakte auf. Diese beeinträchtigen das Ergebnis und können u. U. eine scheinbare Verschiebung der TM-Werte zu höheren (oder niedrigeren) Temperaturen bewirken. Manche Proteine und Pufferbedingungen sind nicht kompatibel mit Fluoreszenzverfahren. In diesem Fall kann die Berechnung der TM-Unterschiede sehr schwierig sein. Und schließlich können mit Fluoreszenz- oder Spektroskopieverfahren nicht die kalorimetrische Enthalpie und andere thermodynamische Parameter bestimmt werden. Die DSC hingegen kann alle diese Informationen liefern und stellt damit einen Thermostabilitätsassay dar.
Die DSC wird in der biopharmazeutischen Industrie als maßgeblicher Thermostabilitätsassay erachtet, weil das Verfahren folgende Vorteile hat:
Es misst die mit der Proteinentfaltung einhergehenden Wärmeänderungen.
Es ermöglicht eine direkte Messung der Proteinentfaltung, ohne dass eine Markierung, eine Sonde oder ein Tag notwendig ist. Daher gibt es hier keine Detektionsartefakte, wie sie bei Fluoreszenzassays oder anderen spektroskopischen Assays häufig auftreten.
Es misst native Proteine in Lösung und kann mit nahezu allen Puffern und Additiven eingesetzt werden, die in der Aufreinigung und Formulierung von Biopharmaka üblich sind. Manche dieser Puffer und Additive sind nicht kompatibel mit Fluoreszenz- oder Spektroskopieverfahren.
Es weist einen einfachen Versuchsaufbau auf.
Es weist eine hochpräzise Temperaturregelung mit einem Arbeitstemperaturbereich bis 130 °C auf, sodass es auch Übergänge mit sehr hohen TM-Werten detektieren kann. Bei anderen Assays zur TM-Bestimmung können die Proben nur bis auf 100 °C (oder weniger) erhitzt werden.
Es handelt sich um einen sogenannten Forced-Degradation-Assay, bei dem das Protein nicht vor der Analyse in Pufferlösung gelagert werden muss. Für SEC-HPLC und DLS müssen die Proben häufig in Puffern bei erhöhten Temperaturen inkubiert werden, um Änderungen der Konformation nachweisen zu können.
Es verfügt über eine einfache Datenausgabe und eine integrierte Datenanalysesoftware.
Es kann zur Auflösung separater Entfaltungsübergänge und zur Charakterisierung von Multidomänenproteinen, Proteinkomplexen und einfachen Einzeldomänenproteinen eingesetzt werden.
Es ist aussagekräftig und liefert thermodynamische Daten zusätzlich zur Bestimmung der konformationellen Stabilität und der TM-Werte.
Es kann als Primärassay zur Charakterisierung der thermischen Stabilität von Biotherapeutika dienen. Zudem kann es auch zusammen mit anderen, orthogonalen oder komplementären biophysikalischen Screening-Methoden sowie zur Validierung anderer Daten verwendet werden.
Es ist in einer automatisierten Version mit hohem Durchsatz erhältlich (MicroCal VP-Capillary DSC Automated), um schnelle Untersuchungen der thermischen Stabilität durchzuführen.
Biopharmazeutische Vergleichbarkeit
Monoklonale Antikörper und andere biopharmazeutische Wirkstoffe sind komplexe Proteine, die von Säuger- oder Bakterienzellen exprimiert werden. Jedes Protein ist einzigartig. Um es zu einem Wirkstoff weiterzuentwickeln, bedarf es umfangreicher Forschung, Charakterisierung und Optimierung. Im Rahmen der Entdeckung und frühen Entwicklung werden die gewünschten Eigenschaften des Proteinwirkstoffs definiert, zu denen Reinheit, Wirksamkeit und Dosierung gehören. Zur Untersuchung dieser Eigenschaften werden biochemische, biophysikalische und biologische Assays entwickelt und während der Produktentwicklung laufend optimiert.
Für jeden Prozess wird eine detaillierte Proteincharakterisierung und HOS-Analyse (Stabilitätsanalyse eingeschlossen) durchgeführt, um die kritischen Qualitätseigenschaften des Proteins und die kritischen Prozessparameter zu definieren. Anhand letzterer müssen die Prozesse über den gesamten Produktlebenszyklus hinweg kontrolliert und validiert werden. Beim QbD-Ansatz (Quality by Design) für die biopharmazeutische Entwicklung sind die kritischen Qualitätseigenschaften und die Stabilitätscharakterisierung Teil der Bewertung des Arzneimittels bei der Verfahrensentwicklung und der Produktionsunterstützung.[16,17]
Während der Verarbeitung eines Proteinwirkstoffs ist das Protein unterschiedlichen Bedingungen ausgesetzt. Hierzu gehören:
Verschiedene Lösungen mit unterschiedlichen Puffern, pH-Werten und Salzen
Formulierungsadditive (Hilfsstoffe)
Verschiedene Proteinkonzentrationen
Wiederholte Gefrier- und Auftauzyklen, erhöhte Drücke und Mischen/Schütteln
Unterschiedliche Materialoberflächen, mit denen das Protein in Kontakt kommt (Pumpen, Ultrafiltrations-/Diafiltrationsmembranen, Chromatographiemedien usw.)
Exposition gegenüber Oxidantien, Proteasen, Zellkulturmedien, Produktvarianten usw.
Diese Bedingungen können die Kräfte und Wechselwirkungen stören, die ein Protein in seiner nativen (gefalteten) Konformation halten, was zu einem denaturierten oder inaktiven Protein führt. Es kann auch eine chemische Denaturierung durch Oxidation, Deamidierung, Änderungen der Glykosylierung oder andere posttranslationale Modifikation eintreten.
Denaturierte Proteine bilden häufig Aggregate. Dies stellt in der biopharmazeutischen Entwicklung ein wesentliches Problem dar. Aggregate sind Zusammenlagerungen von Proteinmonomeren. Die Aggregatbildung kann reversibel oder irreversibel sein. Der Größenbereich reicht von Proteindimeren bis zu großen Partikeln, die mit bloßem Auge sichtbar sind. Durch die Proteinaggregation werden zumindest die Wirksamkeit und Wirkstärke des Arzneimittels verringert und die Herstellungskosten erhöht. Mehr Anlass zur Besorgnis gibt das Potenzial von Proteinaggregaten und -partikeln, immunogene Reaktionen auszulösen, die im Extremfall zum Tod des Patienten führen können. Aufgrund dieses Problems ist die Proteinaggregation in der biopharmazeutischen Industrie Gegenstand einer genauen Überprüfung. Diese umfasst die Partikel-/Aggregationserkennung, die Quantifizierung und Charakterisierung in der Produktion und Formulierung sowie die Suche nach Lösungen zur Reduzierung oder Eliminierung der Proteinaggregation bei biopharmazeutischen Produkten.
Es ist wichtig nachzuweisen, dass der hergestellte Proteinwirkstoff hinsichtlich Struktur, Stabilität, Größenverteilung und biochemischen und funktionellen Assays vergleichbar ist mit:
dem gleichen Protein, das in früheren Chargen hergestellt wurde, sowie mit der Referenzproteincharge.
dem gleichen Protein, das an unterschiedlichen Standorten hergestellt wurde.
dem gleichen Protein, das mit einem modifizierten Upstream- und Downstream-Prozess hergestellt wurde.
dem Scale-up des Upstream- und Downstream-Prozesses.
dem gleichen Protein in einer anderen Formulierung.
Es werden biochemische, biophysikalische, funktionelle und HOS-Assays zum Nachweis der Komparabilität (oder Biokomparabilität) entwickelt. Mit ihnen kann gezeigt werden, dass das hergestellte Proteinprodukt hinsichtlich der vorgesehenen kritischen Qualitätseigenschaften sehr ähnlich zu einem Referenzprotein ist. Es ist von entscheidender Bedeutung, dass die HOS der Proteinwirkstoffkandidaten in jeder Phase der Entwicklung sorgfältig analysiert wird.
Änderungen am Herstellungsprozess eines Biopharmazeutikums können sowohl während der Prozessentwicklung als auch nach der Arzneimittelzulassung erfolgen. Zu den Gründen für solche Änderungen gehören:
Verbesserung des Herstellungsprozesses (z. B. Produktausbeute, Kostenreduzierung)
Maßstabsvergrößerung der Produktion
Änderung des Produktionsstandorts
Rechtliche und regulatorische Änderungen
Verbesserung der Produktstabilität
Werden wesentliche Änderungen am Produktionsprozess vorgenommen, muss eine Vergleichbarkeitsuntersuchung durchgeführt werden, um die Auswirkungen der Änderungen auf die kritischen Qualitätseigenschaften, die Sicherheit und die Wirksamkeit des biopharmazeutischen Produkts zu beurteilen. Wenn die Vergleichbarkeit nachgewiesen wurde, bedeutet dies nicht zwangsläufig, dass die kritischen Qualitätseigenschaften des Produkts vor und nach der Änderung identisch sind. Sie sind jedoch sehr ähnlich. Produktionsänderungen dürfen keine negativen Auswirkungen auf die Produktqualität haben.
Die Vergleichbarkeit ist bei Proteintherapeutika ein entscheidendes Kriterium, das von mehreren internationalen Zulassungsbehörden berücksichtigt wird.[18,19,20] Um die Vergleichbarkeit von Proteinwirkstoffen zu beurteilen, ist keine einzelne analytische Methode allein ausreichend. Für die HOS- und Vergleichbarkeitsanalyse werden u. a. eingesetzt: DSC, DLS, Fluoreszenz, CD, AUC, SEC, Raman-Spektroskopie, Nanopartikel-Tracking-Analyse (NanoSight von Malvern Instruments), Resonanzmassenmessung (Archimedes von Malvern Instruments) und Mikroskopie. Zudem werden Protein-Bioaktivität und Wirksamkeit mit Bioassays, isothermer Titrationskalorimetrie (ITC) und Oberflächenplasmonenresonanz (SPR) überprüft.
Die von den einzelnen biophysikalischen Verfahren generierten Daten sind spezifisch. Bei gemeinsamer Bewertung liefern sie Strukturinformationen für die HOS-Charakterisierung und Vergleichbarkeitsstudien. Anhand der „HOS-Kartierung“ kann eine gleichbleibende Proteinstruktur gewährleistet werden, wenn z. B. ein Scale-up der Verfahrensentwicklung erfolgt, und eine Chargenvergleichbarkeit, wenn Prozesse an neue Produktionsstandorte verlagert werden oder die Produktion modifiziert wird. Die Informationen aus der HOS-Charakterisierung sind auch für die Evaluierung von Biosimilars von unschätzbarem Wert.
Anzumerken ist, dass sich die Überprüfung und Herstellung der Vergleichbarkeit während eines Produktionsprozesses (oder dessen Änderung) von den Überprüfungen in der Entwicklung eines Biosimilar-Arzneimittels unterscheiden: Die Biosimilar-Entwicklung ist ein weitaus komplexerer Prozess. Biosimilars werden in der Regel nicht von demselben Unternehmen hergestellt, das den Innovator (auch Parental- oder Referenzprodukt genannt) entwickelt hat. Daher muss in der Biosimilar-Entwicklung Reverse Engineering eingesetzt werden, um neue Upstream- und Downstream-Prozesse zu entwickeln. Der Biosimilar-Hersteller muss die Produktqualität (Biosimilarität) eng an die Spezifikationen des kommerziellen Innovatorprodukts halten. Hierzu benötigt er mehr biophysikalische und biochemische Nachweise, um die Biosimilarität zu belegen, als für den Beleg der Vergleichbarkeit bei der Herstellung typischerweise benötigt werden.
Die DSC liefert Informationen zur thermischen Stabilität eines Proteins unter verschiedenen Lösungsmittelbedingungen. Zudem sind die DSC-Ergebnisse reproduzierbar (Abbildung 4). Daher wird die DSC häufig in Vergleichbarkeitsstudien eingesetzt, um nachzuweisen, dass durch den Prozess keine Veränderung der Produktstabilität bewirkt wird und dass die hergestellten Produkte aus verschiedenen Chargen und/oder Produktionsstandorten sehr ähnlich sind.
Jiang und Nahri[21] überprüften mehrere biophysikalische Methoden zur Bewertung der Vergleichbarkeit bei der Prozessentwicklung und Herstellung. Sie beschrieben die DSC als eines der biophysikalischen Charakterisierungswerkzeuge für Vergleichbarkeitsstudien. Diese Studien betrafen ein monoklonales Antikörperarzneimittel, das von zwei unterschiedlichen Zelllinien exprimiert wird (Zelllinien 1 und 2), wobei zusätzlich eine Modifikation der Produktionsprozesse erfolgte (Prozesse 2pA und 2pB). Die drei resultierenden Proteinprodukte wurden mit den folgenden Verfahren beurteilt: FTIR (zur Bestimmung der Sekundärstruktur), Nah-UV-CD und intrinsische Fluoreszenz (zum Vergleich der Tertiärstruktur), Fluoreszenz und ANS-Bindung (zum Vergleich der Oberflächenhydrophobizität), DLS (zum Vergleich der hydrodynamischen Eigenschaften und der Größenverteilung der Proteine) und DSC (zum Vergleich der thermischen Stabilität und Löslichkeit).
Die Ergebnisse aus dieser Reihe von biophysikalischen Assays ließen darauf schließen, dass die gesamten Sekundär- und Tertiärstrukturen der drei Proben ähnlich waren. Die DSC-Ergebnisse zeigten, dass die thermische Stabilität des Proteins der Zelllinie 2pB gegenüber den Proben der Zelllinien 2pA und 1 signifikant erhöht war. Zudem schienen die Proben der Zelllinien 2pA und 1 laut DSC heterogener zu sein. Bei Antikörpern können üblicherweise 2 bis 3 Übergänge beobachtet werden (Abbildung 2 zeigt ein Beispiel eines „typischen“ DSC-Thermogramms eines Antikörpers). Die DSC-Analyse dieser Proben ergab eine höhere Anzahl überlappender thermischer Übergänge und weist damit auf eine Heterogenität der Proben hin. Aus den DSC-Ergebnissen geht hervor, dass die Homogenität und Stabilität des gewünschten monoklonalen Antikörpers durch die Prozessmodifikation verbessert wurden. Aus den Vergleichbarkeitsdaten aus den in dem Artikel beschriebenen biophysikalischen, biochemischen und funktionellen Assays wurde geschlossen, dass die Zelllinie 2 und der Prozess 2pB das stabilere Protein produzierten.[21]
In einem zweiten Beispiel von Jiang und Nahri[21] wurde die DSC eingesetzt, um die Vergleichbarkeit eines Proteinprodukts zu untersuchen, das an unterschiedlichen Produktionsstandorten hergestellt wurde. Protein Y bestand aus zwei monomeren Polypeptidketten mit einer Disulfidbrücke am Fc-Bereich des Moleküls. Im Verlauf der Entwicklung wurde Protein Y an unterschiedlichen Produktionsstandorten hergestellt. Es sollte verifiziert werden, dass das Protein aus diesen unterschiedlichen Chargen hinsichtlich nativer Konformation, Sekundär- und Tertiärstrukturen, thermischer Stabilität und Größenverteilung vergleichbar war. Hierzu wurden vier verschiedene Proben von Protein Y – drei an unterschiedlichen Standorten hergestellte Proben und eine Referenzproteinprobe – mittels FTIR, Nah-UV-CD, Fluoreszenz, DSC und AUC analysiert.
Die DSC-Thermogramme des Referenzstandards und der 3 Proben von unterschiedlichen Produktionsstandorten wiesen jeweils zwei thermische Übergänge auf. Alle vier DSC-Profile waren innerhalb der Messtoleranzen identisch. Daraus ließ sich schließen, dass hinsichtlich der thermischen Stabilität der Proteinproben keine Unterschiede existierten und dass alle Proben in der nativen Konformation gefaltet vorlagen. Die kombinierten Ergebnisse aus der biophysikalischen Charakterisierung, einschließlich jener der DSC, zeigten, dass alle vier Proben von Protein Y vergleichbar waren, die entsprechende Sekundär- und Tertiärstruktur aufwiesen und homogen waren.[21]
Bei nicht identischen DSC-Thermogrammen hätte man auf eine Änderung der posttranslationalen Modifikation oder auf eine chemische Denaturierung des Proteins rückschließen können, die mit dem Prozess oder der Formulierung in Zusammenhang steht. Arthur et al.[22] demonstrierten die Empfindlichkeit der DSC bei der Erkennung von stabilitätsrelevanten HOS-Veränderungen durch Oxidation, einem häufigen chemischen Degradationsweg. Erfährt ein biopharmazeutisches Produkt bei der Aufreinigung oder der Lagerung in der endgültigen Formulierung eine Oxidation, kann sich dies negativ auf die Wirksamkeit auswirken und auch die Wahrscheinlichkeit einer Proteinaggregation erhöhen. In dieser Studie wurden Proteinprodukte aus drei strukturellen Klassen bei verschiedenen Oxidationsgraden untersucht. Jedes Protein zeigte eine lineare Abnahme des TM-Werts als Funktion der Methioninoxidation. Zudem beobachteten die Autoren Unterschiede bei der Veränderungsrate der TM-Werte sowie Unterschiede bei der Stabilität der Domänen-TM-Werte – quer über alle strukturellen Klassen und innerhalb derselben.[22] Im Gegensatz dazu waren Nah-UV-CD und Fluoreszenzspektroskopie gegenüber oxidationsinduzierten Konformationsänderungen weitaus weniger empfindlich. Im Vergleich zu diesen spektroskopischen Verfahren ist die DSC somit ein geeigneteres strukturelles Charakterisierungsverfahren zur Überwachung der Proteinoxidation.[22] Bei einem Protein in der Studie (IgG2B) wurden mittels DSC Veränderungen des TM-Werts detektiert, die einem Abfall der relativen Wirkstärke vorangingen. Dies zeigt, dass die DSC ein Frühindikator einer verminderten Antigenbindung ist. Mittels Massenspektrometrie (MS) konnten Veränderungen des oxidierten Methionins nachgewiesen werden. Diese traten bei Oxidationsgraden auf, die zu niedrig waren, um messbare konformationelle oder funktionelle Auswirkungen zu verursachen. Anhand der TM-Verschiebungen aus der DSC und der Ergebnisse der MS- und Wirkstärkemessungen kann der Zusammenhang zwischen einer Modifikation der Primärstruktur, Veränderungen der HOS und der konformationellen Stabilität und funktionellen Auswirkungen zuverlässig charakterisiert werden.
Morar-Mitrica et al.[12] beschreiben mehrere Fallbeispiele für den Einsatz der DSC in HOS- und Vergleichbarkeitsanalysen von Biopharmazeutika. Diese Beispiele umfassen die Charakterisierung eines oxidierten monoklonalen Antikörpers anhand feststellbarer TM-Verschiebungen (ähnlich zu den in [22] beschriebenen), die Verwendung reproduzierbarer DSC-Thermogrammprofile und der Werte TM, T1/2, Tonset und ∆H für Komparabilitätsstudien von Biopharmazeutika sowie die erweiterte DSC-Charakterisierung glykosylierter monoklonaler Antikörper mit unterschiedlichen Graden der Glykosylierung (einer häufigen posttranslationalen Modifikation).
Shahrokl et al.[23] erörtern den Nutzen der DSC in Vergleichbarkeitsstudien im Rahmen des Zulassungsantrags (Biological License Application, BLA). Dabei erwähnen sie die einfache Anwendung der DSC und ihre einfache Datenausgabe. Die Autoren beschreiben den Einsatz der DSC für Komparabilitätsstudien bei einem 51-kDa-Glykoprotein vor und nach einer Änderung in seinem Herstellungsprozess. Die Prozessänderung erfolgte, um die Produktausbeute zu steigern und die aus dem Zellkulturmedium stammenden, aus Tieren gewonnenen Materialien zu entfernen. Die Autoren untersuchten mittels DSC drei Chargen des Glykoproteins, die mit den unterschiedlichen Produktionsprozessen hergestellt wurden. Sie stellten fest, dass die DSC-Thermogramme deckungsgleich waren und einen einzelnen Übergang bei 60,7 °C +/- 0,1 °C aufwiesen. Die DSC-Ergebnisse zeigten, dass die konformationelle Stabilität des Glykoproteins durch die Produktionsänderung nicht beeinträchtigt wurde.[23]
Lubiniecki et al.[24] untersuchten die Auswirkungen von Produktionsänderungen auf die Antikörperstruktur und -funktion im Zuge der Produktentwicklung. Hierzu wurden drei Komparabilitätsstudien für zwei verschiedene monoklonale IgG1-Antikörperkandidaten (Antikörper A und Antikörper B) durchgeführt. Zu den eingesetzten biochemischen und biophysikalischen Analysewerkzeugen gehörte bei zwei Komparabilitätsstudien auch die DSC. In einer der Studien wurden das Scale-up und die Verlagerung an den Produktionsstandort untersucht, einhergehend mit der Änderung von einer lyophilisierten zu einer flüssigen Darreichungsform. Die DSC-Thermogramme der lyophilisierten und flüssigen Formulierungen für Antikörper A und Antikörper B zeigten eine gute Übereinstimmung.[24] Die DSC in Kombination mit MS, CD, SEC und anderen Assays zeigte, dass die Struktur und Funktion der Antikörper durch die Produktionsänderung nicht beeinträchtigt wurden.
Bei der dritten Komparabilitätsstudie wurde die Antikörper-Flüssigformulierung in Fertigspritzen und Injektionsfläschchen untersucht.[24] Die Ergebnisse von DSC, CD, SEC und anderen Assays deuteten auf ähnliche Molekularstruktur, biologische Aktivität und Degradationsprofile hin. Die einzige bemerkenswerte Ausnahme war eine geringfügige, aber signifikante Erhöhung der Anzahl subvisibler Partikel in Fertigspritzen.[24]
Ein Biosimilar-Produkt (auch Biogenerikum oder Nachahmerbiologikum genannt) ist ein Biopharmazeutikum, das von einer Zulassungsbehörde aufgrund seiner hohen Ähnlichkeit zu einem bereits zugelassenen biologischen Produkt zugelassen wird. Letzteres wird als Referenzprodukt (auch Ausgangs- oder Innovatorprodukt) bezeichnet. Biosimilars sind seit einigen Jahren weltweit erhältlich.[25-29] Zum Zeitpunkt der Erstellung dieses Whitepapers (Oktober 2016) hatten die US-amerikanische FDA vier Biosimilars und die EMEA 19 Biosimilars zugelassen.
Die Entwicklung eines Biosimilars unterscheidet sich jedoch von der Entwicklung eines generischen niedermolekularen Arzneimittels. Niedermolekulare Wirkstoffe sind Chemikalien, und ihre Synthese ist ein kontrollierter Prozess. Da Biopharmazeutika in der Regel Proteine sind, weisen sie einen bestimmten Anteil von Proteinvariationen auf, selbst zwischen verschiedenen Chargen des gleichen Produkts. Dies ist auf die inhärente Variabilität des biologischen Expressionssystems und des Produktionsprozesses zurückzuführen. Diese Variationen können Unterschiede der posttranslationalen Modifikation und HOS umfassen. Aufgrund der hohen Molekulargewichte, komplexen Strukturen und natürlichen Variationen von Proteinen sind Biosimilars nicht so einfach herzustellen wie Generika. Ein Biosimilar ist kein exaktes Duplikat des Referenzprodukts. Aus diesem Grund wird es üblicherweise als „ähnlich“ oder „sehr ähnlich“ zum Referenzprodukt charakterisiert.
Die Entwicklung eines Biosimilars beinhaltet physikochemische, analytische und funktionale Vergleiche des Referenzproteins mit dem Biosimilar. Diese Assays werden durch nichtklinische und klinische Vergleichsdaten ergänzt, um eine gleichwertige Wirksamkeit und Sicherheit zu belegen. Vor der Zulassung muss nachgewiesen werden, dass das Biosimilar gegenüber dem Referenzprodukt keine klinisch relevanten Unterschiede hinsichtlich Sicherheit und Wirksamkeit aufweist. Bei Biosimilar-Produkten sind nur geringfügige Unterschiede bei klinisch inaktiven Komponenten zulässig. Das Biosimilar muss auf exakt die gleiche Art funktionieren wie das Referenzprodukt – für dieselben zugelassenen Indikationen.
Die Zulassungsbehörden bewerten Biosimilars nach dem Grad der Ähnlichkeit zu ihren Referenzprodukten. Aufgrund der Komplexität von Biopharmazeutika ist es unwahrscheinlich, dass zwei verschiedene Hersteller zwei identische Produkte produzieren, auch wenn identische Wirts-Expressionssysteme, Prozesse und äquivalente Technologien genutzt werden. Daher müssen sich die Biosimilar-Hersteller auf Komparabilitätsstudien und HOS-Analysen verlassen, wie vorstehend erörtert.[30,31] Der Bedarf für mehr analytische Informationen und der Wunsch nach verkürzten Zeitplänen bei der Entwicklung von Biosimilars erfordern den Nachweis der Vergleichbarkeit mit dem Referenzmolekül in jeder Phase, insbesondere bei der Herstellung.
Biosimilars müssen durch Reverse Engineering so eng wie möglich an das Referenzprodukt angepasst werden. Die analytische Charakterisierung eines Biosimilars umfasst die Beurteilung der Primärstruktur, der höheren Ordnungsstruktur (Sekundär-, Tertiär- und Quartärstruktur) und der biologischen Aktivität sowie die Analyse von Produkt- und Prozessverunreinigungen. Die DSC wird oft als biophysikalischer HOS-Assay eingesetzt, um zu zeigen, dass ein Biosimilar im Vergleich zum Referenzprodukt einen sehr ähnlichen „Fingerprint“ im DSC-Profil aufweist sowie ähnliche TM- und Tonset-Werte und andere thermodynamische Parameter.
Bei drei der vier von der US-amerikanischen FDA zugelassenen Biosimilars (Stand Oktober 2016) gehörte die DSC zu den HOS-Assays im FDA-Antrag:
Erelzi (von Sandoz, Inc.) ist ein Biosimilar von Amgens Enbrel (Etanercept).[32]
Inflectra (von Celltrion), in anderen Märkten Remsima, ist ein Biosimilar von Janssens Remicade (Infliximab).[33,34]
Amjevita (von Amgen) ist ein Biosimilar von Abbvies Humira (Adalimumab).[35,36]
Sinha-Datta et al.[37] demonstrierten den Einsatz der DSC (MicroCal VP-Capillary DSC) und der Oberflächenplasmonenresonanz (SPR) als analytische Werkzeuge für den Vergleich zweier therapeutischer monoklonaler Antikörper (mAb1-i und mAb2-i) mit ihren Biosimilars (mAb1-B und mAb2-B1, B2, B3) hinsichtlich thermischer Stabilität, Kinetik und Affinität. Sie stellten fest, dass die Biosimilars ihren Referenzproben hinsichtlich der Biofunktionalität (SPR-Ergebnisse) sehr ähnlich waren. Mit der DSC wurde die Biosimilarität zwischen den Ausgangs- und Biosimilar-Produkten zusätzlich bestätigt. Die DSC-Analyse ergab eine hohe strukturelle Ähnlichkeit zwischen dem parentalen Antikörper und dem Biosimilar-Antikörper. Der primäre TM-Wert betrug 84,1 °C (für mAb1) bzw. 72,8 °C (für mAb2).
Die in diesem Whitepaper vorgestellten Informationen belegen die Bedeutung und den Nutzen der DSC als biophysikalischer Stabilitäts- und HOS-Assay für biopharmazeutische Vergleichbarkeitsstudien und die Charakterisierung von Biosimilars. Anhand der Ergebnisse der DSC und anderer biophysikalischer und biochemischer Assays können biopharmazeutische Unternehmen fundierte Entscheidungen hinsichtlich der Proteinstabilität und Vergleichbarkeit in der Produktion treffen. So kann gewährleistet werden, dass alle Proteinchargen der Referenzcharge sehr ähnlich sind und dass eventuelle Prozess- oder Produktionsänderungen nicht die konformationelle Proteinstabilität beeinträchtigen. Die DSC gehört auch zu den HOS-Assays in der Entwicklung von Biosimilars, um nachweisen zu können, dass das Biosimilar dem Innovator sehr ähnlich ist.
Analytical Techniques for Biopharmaceutical Development, R. Rodriguez-Diaz, T. Wehr, S. Tuck (Hrsg.), Taylor & Francis, New York, NY, USA (2005).
Biophysical Characterization of Proteins in Developing Biopharmaceuticals, D.J. Houde, S.A. Berkowitz (Hrsg.), Elsevier, Amsterdam, Niederlande (2015).
Biophysical Methods for Biotherapeutics: Discovery and Development Applications, T.K. Das (Hrsg.) John Wiley & Sons, Hoboken, NJ, USA (2014).
Biophysics for Therapeutic Protein Development, L.O. Nahri (Hrsg.), Springer, New York, NY, USA (2013).
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 1. Monoclonal Antibody Therapeutics: Structure, Function, and Regulatory Space, J.E. Schiel, D.D. Davis, O.V. Borisov (Hrsg.), ACS Symposium Series Vol 1176 (2014). doi: 10.1021/bk-2014-1176.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 2. Biopharmaceutical Characterization: The NISTmAb Case Study, J.E. Schiel, D.D. Davis, O.V. Borisov (Hrsg.), ACS Symposium Series Vol 1201 (2015). doi: 10.1021/bk-2015-1201.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 3. Defining the Next Generation of Analytical and Biophysical Techniques, J.E. Schiel, D.D. Davis, O.V. Borisov (Hrsg.), ACS Symposium Series Vol 1202 (2015). doi: 10.1021/bk-2015-1202.
1. Gokarn, Y., Agarwal, S., Arthur, K., et al., Biophysical Techniques for Characterizing the Higher Order Structure and Interactions of Monoclonal Antibodies, in: State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 2. Biopharmaceutical Characterization: The NISTmAb Case Study. J.E. Schiel, D.D. Davis, O.V. Borisov (Hrsg.), ACS Symposium Series Vol 1201, American Chemical Society, Washington DC, USA, 285-327 (2015).
2. Cooper, A., Nutley, M. A., Wadood, A., Differential Scanning Calorimetry, in: Protein-Ligand Interactions: Hydrodynamics and Calorimetry, A Practical Approach, S.E. Harding, B.Z. Choudry (Hrsg). Oxford University Press, Oxford, UK, 287-318 (2001).
3. Malvern Instruments Whitepaper „Differential Scanning Calorimetry (DSC) Theory and Practice“ http://www.malvern.com/en/support/resourcecenter/Whitepapers/WP140701-dsc-theory-and-practice.aspx.
4. Bruylants, G., Wouters, J., Michaux, C., Current Med. Chem. 12, 2011-2020 (2005). doi: 10.2174/0929867054546564.
5. Jelesarov, I., Bosshard, H.R., J. Mol. Recognit. 12, 3-18 (1999). doi: 10.1002/ (SICI)1099 1352(199901/02)12:1<3:AID-JMR441>3.0.CO; 2-6.
6. Choi, M.H., Prenner, E.J., J. Pharm. Bioallied Sci. 3, 39-59 (2011). doi: 10.4103/0975-7406.76463.
7. Johnson, C.M., Arch. Biochem. Biophys. 531, 100-109 (2013). doi: 10.1016/j.abb.2012.09.008.
8. Plotnikov, V., Rochalski, A., Brandts, M., Brandts, J.F., Williston, S., Frasca, V., Lin, L.N., Assay Drug Devel. Technol. 1, 83-90 (2004). doi:10.1089/154065802761001338.
9. www.malvern.com
10. Demarest, S.J., Frasca. V., Differential Scanning Calorimetry in the Biopharmaceutical Sciences, in: Biophysical Characterization of Proteins in Developing Biopharmaceuticals, D.J. Houde, S.A. Berkowitz (Hrsg.), Elsevier, Amsterdam, Niederlande, 287-306 (2015).
11. Remmele, R.L., Microcalorimetric Approaches to Biopharmaceutical Development, in: Analytical Techniques for Biopharmaceutical Development, R. Rodriguez-Diaz, T. Wehr, S. Tuck (Hrsg.), Taylor & Francis, New York, NY, USA, 327-381 (2005).
12. Morar-Mitrica, S., Nesta, D., Crofts, G., BioPharm Asia 2, 46-55 (2013).
13. Kirkitadze, M., Hu, J., Tang, M., Carpick, B., Pharm. Bioprocess. 2, 491-498 (2014). doi: 10.4155/PBP.14.27.
14. Wen, J., Arthur, K., Chemmalil, L., Muzammil, S., Gabrielson, J., Jiang, Y., J. Pharm. Sci. 101,955-964 (2012). doi: 10.1002/jps.22820.
15. Gabrielson, J.P., Weiss, W.F., J. Pharm. Sci. 104, 1240-1245 (2015). doi: 10.1002/jps24393.
16. Cooney, B., Jones, S.D., Levine, H., BioProcess Int. 14(6), 28-35 (2016). http://www.bioprocessintl.com/analytical/upstream-development/quality-by-design-for-monoclonal-antibodies-part-1-establishing-the-foundations-for-processdevelopment/
17. Cooney, B., Jones, S.D., Levine, H., BioProcess Int. 14(8), 24-33 (2016). http://www.bioprocessintl.com/2016/quality-design-monoclonal-antibodies-part-2-process-design-space-control-strategies/
18. http://www.who.int/biologicals/biotherapeutics/rDNA_DB_final_19_Nov_2013.pdf
19 http://www.ema.europa.eu/docs/en_GB/document_library/Scientific_guideline/2009/09/WC500003935.pdf
20. http://www.fda.gov/downloads/Drugs/GuidanceComplianceRegulatoryInformation/Guidances/UCM496611.pdf
21. Jiang, Y., Nahri, L.O., J. Am. Pharm. Rev. 9, 34-43 (2006).
22. Arthur, K.K., Dinh, N. Gabrielson, J. P., J. Pharm. Sci. 104, 1544-1554 (2015). doi: 10.1002/jps.24313.
23. Shahrokh, Z., Salamat-Miller, N., Thomas, J.J., Biophysical Analyses Suitable for Chemistry, Manufacturing, and Control Sections of the Biologic License Application (BLA), in: Biophysical Methods for Biotherapeutics: Discovery and Development Applications, T.K. Das (Hrsg.) John Wiley & Sons, Hoboken; NJ; USA; 317-353 (2014).
24. Lubiniecki, A., Volkin, D.B., Federici, M, et al., Biologicals39, 9-22 (2011). doi: 10.1016/j.biologicals.2010.08.004.
25.http://www.fda.gov/Drugs/DevelopmentApprovalProcess/HowDrugsareDevelopedandApproved/ApprovalApplications/TherapeuticBiologicApplications/Biosimilars/
26.http://www.ema.europa.eu/docs/en_GB/document_library/Scientific_guideline/2015/01/WC500180219.pdf
27.http://www.who.int/biologicals/areas/biological_therapeutics/BIOTHERAPEUTICS_FOR_WEB_22APRIL2010.pdf
28 Bas, T.G., Oliu Castillo, C., Biomed Res Int. 2016, 5910403 (2016). doi: 10.1155/2016/5910403.
29. Tsuruta, L.R., Lopes dos Santos, M., Moro, A.M., Biotechnol Prog. 31, 1139-1149 (2015). doi: 10.1002/btpr.2066.
30 Kálmán-Szekeres, Z., Olajos, M., Ganzler, K., J. Pharm. Biomed. Anal. 69, 185-195 (2012). doi: 10.1016/j.jpba.2012.04.037.
31 Berkowitz, S.A., Engen, J.R., Mazzeo, J.R., Jones, G.B., Nat. Rev. Drug Discov. 11, 527-540 (2012). doi: 10.1038/nrd3746.
32.http://www.fda.gov/downloads/AdvisoryCommittees/CommitteesMeetingMaterials/Drugs/ArthritisAdvisoryCommittee/UCM510493.pdf
33.http://www.fda.gov/downloads/AdvisoryCommittees/CommitteesMeetingMaterials/Drugs/ArthritisAdvisoryCommittee/UCM484860.pdf
34. Jung, S.K., Lee, K. H., Jeon, J. W., et al., MAbs 6, 1163-1177 (2014). doi: 10.4161/mabs.32221.
35.http://www.fda.gov/downloads/AdvisoryCommittees/CommitteesMeetingMaterials/Drugs/ArthritisAdvisoryCommittee/UCM510293.pdf
36. Liu, J., Eris, T., Li, C., Cao, S., Kuhns, S., BioDrugs 30, 321-338 (2016). doi: 10.1007/s40259-016-0184-3.
37. Sinha-Datta, U., Khan, S., Wadgaonkar, D., Biosimilars5, 83-91 (2015): doi: 10.2147/BS.S85537 https://www.dovepress.com/label-free-interaction-analysis-as-a-tool-to-demonstrate-biosimilarity-peer-reviewed-article-BS