Die Stabilität eines biopharmazeutischen Proteins ist entscheidend für den Erfolg oder Misserfolg der Entwicklung eines brauchbaren Wirkstoffs. Die Proteinstabilität spielt eine entscheidende Rolle hinsichtlich Produktion, Herstellung, Formulierung, Langzeit-Lagerfähigkeit, Verabreichung an den Patienten und Wirksamkeit. Hochstabile Proteine neigen weniger zu Problemen während des Herstellungsprozesses, sind kostengünstiger in der Produktion und bleiben mit größerer Wahrscheinlichkeit während Formulierung und Lagerung funktionsfähig, ohne chemische Veränderungen oder Aggregation. Beim QbD-Ansatz (Quality by Design) für die biopharmazeutische Entwicklung ist die Stabilitätscharakterisierung Teil der Bewertung der Entwicklungsfähigkeit und Wirkstofffähigkeit eines Kandidatenmoleküls. Sie wird auch während der Verfahrensentwicklung und Herstellung durchgeführt. Die Stabilitätsdaten fließen zudem in die Charakterisierung der höheren Ordnungsstruktur (HOS) und das Fingerprinting ein. Diese Daten werden zur Unterstützung der Herstellung sowie für biopharmazeutische Vergleichsuntersuchungen und Biosimilaritätsstudien herangezogen. Des Weiteren wird die HOS-Charakterisierung von Proteinen bei den Zulassungsanträgen für neue biopharmazeutische Wirkstoffe und Biosimilars zunehmend vorausgesetzt.
Aufgrund des komplexen Charakters von Proteinen spielen biophysikalische Werkzeuge bei der vollständigen Charakterisierung eines biopharmazeutischen Produkts eine wichtige Rolle. Zur Bewertung der Proteinstabilität stehen verschiedene biophysikalische Werkzeuge zur Verfügung. Hierzu gehören u. a die CD-Spektroskopie (CD), die dynamische und statische Lichtstreuung (DLS und SLS), die Größenausschlusschromatographie mit Mehrwinkel-Lichtstreudetektoren (SEC-MALS), die Fourier-Transformations-Infrarotspektrometrie (FTIR), die analytische Ultrazentrifugation (AUC), die Größenausschlusschromatographie (SEC), die Differential-Scanning-Fluorimetrie (DSF), die intrinsische Fluoreszenz (IF) und die dynamische Differenzkalorimetrie (DSC).
Wenngleich all diese biophysikalischen Assays in der biopharmazeutischen Entwicklung eine wichtige Rolle spielen, ist die Charakterisierung der thermischen Stabilität mittels DSC von besonderer Bedeutung. Ein Artikel aus dem Jahr 2015 über biophysikalische Methoden zur Charakterisierung der höheren Ordnungsstruktur monoklonaler Antikörper (Gokarn et al.) stellt fest: „Die DSC ist nach wie vor ein konkurrenzloses Verfahren zur Beurteilung der thermodynamischen Stabilität von Proteinen unter gegebenen Pufferbedingungen.“[1]
Dieses Whitepaper soll aufzeigen, wie bei der Auswahl von Wirkstoffkandidaten für die Entwicklung die DSC zur Charakterisierung der thermischen Stabilität von Protein-Biopharmazeutika (v. a. von Antikörpern) eingesetzt wird.
Die DSC ist ein mikrokalorimetrisches Verfahren, mit dem die thermische und konformationelle Stabilität von Proteinen, Nukleinsäuren, Lipiden und anderen Biopolymeren charakterisiert werden kann.[2-7] Die DSC misst die Wärmekapazität als Funktion der Temperatur. Bei den für die Proteincharakterisierung eingesetzten DSC-Geräten, wie sie in diesem Whitepaper beschrieben werden, handelt es sich um Leistungsdifferenzkalorimeter. Dabei wird das Biopolymer in Lösung in eine ortsfeste Probenzelle gegeben, und eine gleichartige Referenzzelle wird mit einer entsprechenden Pufferlösung befüllt. Der für die Probenzelle gemessene Wärmekapazitätswert (Cp) wird mit dem der Referenzzelle verglichen. Während die Temperatur der Zellen erhöht wird, wird der Temperaturunterschied zwischen Referenz- und Probenzelle kontinuierlich gemessen und in Leistungswerte umgewandelt. Die DSC ist ein sogenannter Forced-Degradation-Assay: Das Protein wird einer steigenden Temperatur ausgesetzt, wodurch es beginnt, sich zu entfalten. Dabei nimmt der Cp-Wert des Proteins zu (Abbildung 1).
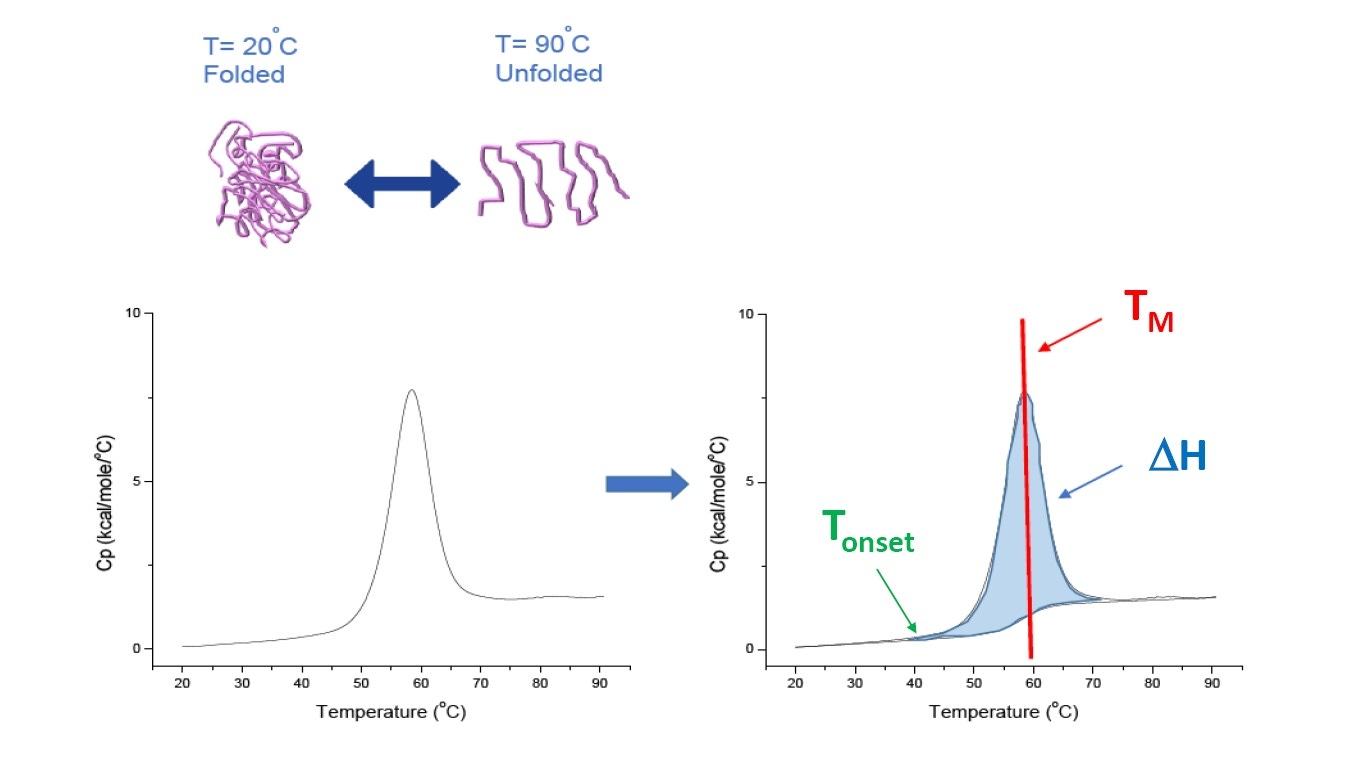
Abbildung 1: Funktionsweise der DSC. Die Wärmekapazität (Cp) ändert sich mit der thermischen Denaturierung des Proteins. Der DSC-Versuch beginnt bei einer Temperatur, bei der das Protein vorwiegend in seiner nativen Konformation gefaltet ist. Bei steigender Temperatur beginnt das Protein ab einem bestimmten Punkt, sich zu entfalten bzw. zu denaturieren (Tonset), und der Cp-Wert nimmt zu. Bei der Temperatur, bei der 50 % des Proteins in der nativen Konformation vorliegen und 50 % denaturiert sind, erreicht der Cp-Wert sein Maximum. Dies ist der Mittelpunkt des thermischen Übergangs, TM. Oberhalb von TM ist das Protein vorwiegend denaturiert, und zum Ende des DSC-Versuchs liegt das Protein vollständig in entfalteter Konformation vor. Zu den Parametern des DSC-Versuchs gehören Tonset, TM und die Entfaltungsenthalpie (∆H).
Die DSC misst die Änderung der Wärmekapazität direkt, ohne dass eine Fluoreszenzmarkierung oder eine andere Markierung oder Sonde benötigt wird. Der Mittelpunkt des thermischen Übergangs (TM) wird auch Schmelz- oder Denaturierungstemperatur genannt. Bei dieser Temperatur liegen 50 % des Proteins in nativer (gefalteter) Konformation und 50 % in denaturierter Konformation vor. Der TM-Wert ist im DSC-Thermogramm als Peak zu erkennen (Abbildung 1).
TM ist ein guter Indikator für die thermische Stabilität – je höher der TM-Wert, desto thermisch stabiler ist das Protein. DSC-Thermogramme von Multidomänenproteinen (wie z. B. von Antikörpern) weisen typischerweise mehrere Peaks auf, sodass mehrere TM-Werte ermittelt werden können (Beispiel siehe Abbildung 2).
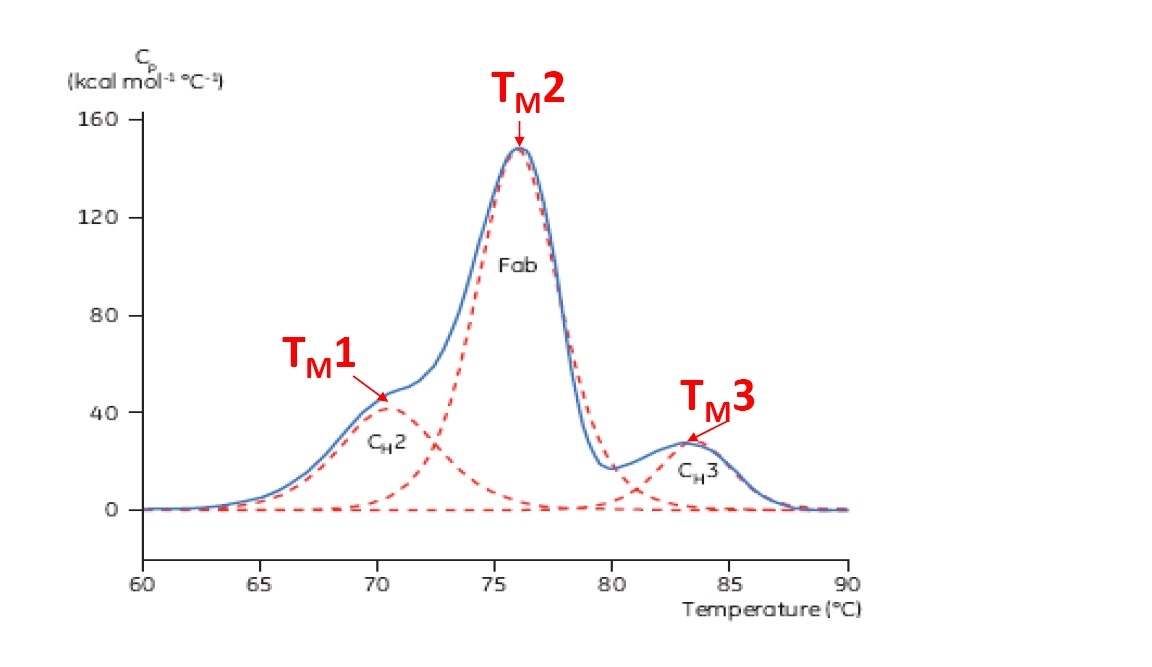
Abbildung 2: Repräsentatives DSC-Thermogramm eines monoklonalen Antikörpers mit Kennzeichnung der CH2-, Fab- und CH3-Domäne. Die gestrichelten roten Linien stellen die dekonvolutierten Peaks der drei einzelnen Domänenübergänge dar. Hierzu sind die jeweiligen TM-Werte gekennzeichnet.
Die DSC liefert auch weitere nützliche Parameter, die zur Charakterisierung und Klassifizierung der Proteinstabilität herangezogen werden können. Hierzu gehört die Entfaltungsenthalpie (∆H), die proportional zur Fläche unter der Kurve ist. Die Proteinentfaltung verläuft endotherm, da eine Energiezufuhr benötigt wird, um die nicht kovalenten sekundären Bindungen aufzubrechen, die das Protein gefaltet halten. Bestimmt werden können mit der DSC zudem die Werte von Tonset (Beginn der Entfaltung), ∆Cp (Wärmekapazitätsänderung bei Entfaltung) und T1/2 (Peakbreite bei halber Peakhöhe, charakteristisch für die Form des Entfaltungsthermogramms). Mittels DSC-Analyse können diese Parameter in beliebiger Kombination bestimmt werden.
Malvern Instruments liefert das System MicroCal VP-Capillary DSC[8,9], ein automatisiertes DSC-Kalorimeter für TM-Untersuchungen und die thermodynamische Charakterisierung von Proteinen und Biopolymeren in Lösung.
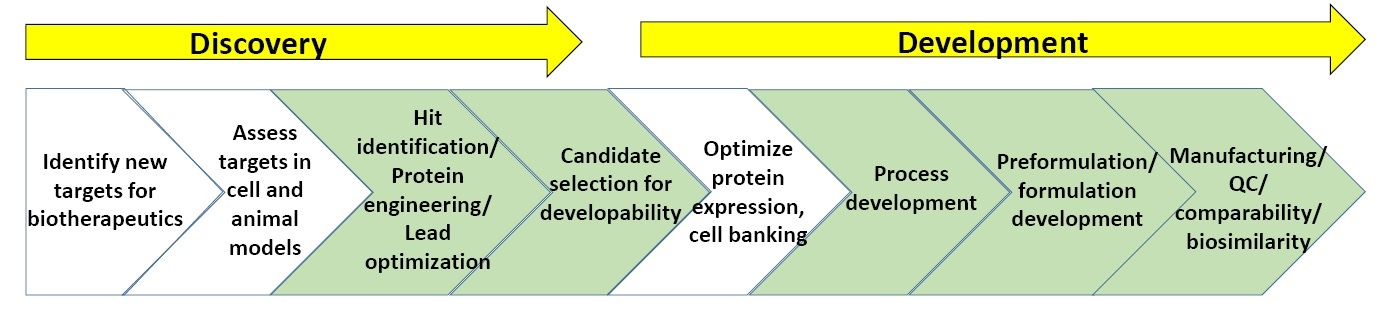
Abbildung 3: Ein allgemeines Schema der Phasen in der Entdeckung und Entwicklung von Biopharmaka.
Abbildung 3 zeigt ein allgemeines Schema der Charakterisierungsschritte in der Entdeckung und Entwicklung von Biopharmaka. In den grün gekennzeichneten Phasen wird die biophysikalische Charakterisierung (Stabilitätsassays eingeschlossen) am häufigsten eingesetzt. Am Ende dieses Whitepapers finden Sie eine Liste empfohlener Literatur zur biopharmazeutischen Forschung und Entwicklung.
Die Reihenfolge der Aminosäuren in einer Polypeptidkette, Primärstruktur (1°) genannt, ist die unterste Ebene der Proteinstruktur. Es ist wichtig, die dreidimensionale Struktur des Proteins zu verstehen und zu charakterisieren. Diese wird auch höhere Ordnungsstruktur (HOS) genannt. Es existieren drei Ebenen der höheren Ordnungsstruktur (HOS) eines Proteins: Die Sekundärstruktur (2°) – sie beschreibt die lokalen Faltungsmuster der Primärstruktur des Proteins, wie die α-Helix, das β-Faltblatt, Schleifen und statistische Knäuel. Die Tertiärstruktur (3°) – sie beschreibt die 3D-Struktur eines Proteins, die aus einer Anordnung von Sekundärstrukturelementen gebildet wird.Die Quartärstruktur (4°) – sie beschreibt Strukturen die infolge der Wechselwirkungen zwischen zwei oder mehreren, identischen oder verschiedenen Polypeptidketten entstehen.
Um ein gewünschtes Biopharmazeutikum zu entwickeln, suchen die Forscher zunächst nach Biomolekülen, die bereits bei der Kandidatenauswahl eine hohe Stabilität zeigen. Anschließend müssen sie u. U. eine weitere Erhöhung der Stabilität durch Protein-Engineering erzeugen. Während der Aufreinigungsphase wird das Protein in der Regel aus Bedingungen entfernt, in denen es stabil, korrekt gefaltet und aktiv ist. Es ist daher wichtig, die richtigen Puffer, Additive, Aufreinigungsverfahren und Lagerungsbedingungen zu verwenden, um das Protein in dieser Phase so stabil wie möglich zu halten.
Wenn Proteinmoleküle Belastungen ausgesetzt werden (Wärme, Chemikalien, pH-Änderungen, Druck, Vermischung und hoher Konzentration), wie sie in der Formulierung und Produktion von Biopharmaka häufig auftreten, kann die Proteinkonformation das denaturierte (entfaltete) Protein begünstigen. Subkutane Formulierungen von Proteinwirkstoffen müssen bei sehr hohen Proteinkonzentrationen (über 100 mg/ml) in den Behältnissen (z. B. Injektionsfläschchen oder Fertigspritzen) stabil und unverändert bleiben – oft über Jahre hinweg. Proteine in Lösung sind zudem empfindlich gegenüber Modifikationen wie Deamidierung und Oxidation, die ebenfalls zu denaturierten, inaktiven Proteinen führen können.
Bei Protein-Biopharmazeutika können eine Denaturation oder andere Modifikationen die Bildung von Aggregaten bewirken. Diese können wiederum dazu führen, dass die Wirksamkeit des Wirkstoffs vermindert oder seine Funktion herabgesetzt ist. Noch kritischer ist, dass die Proteinaggregation mit potenziell lebensbedrohlichen immunogenen Reaktionen des Patienten in Verbindung gebracht wird. Die Verwendung stabiler Proteinwirkstoffe führt zu einer kostengünstigeren Produktion und zu erfolgreicheren, wirksameren und sichereren Arzneimitteln.
Die DSC liefert ein Maß der konformationellen Stabilität eines Moleküls und der Änderungen der Tertiär- und Quartärstruktur, die auftreten, wenn ein Protein thermisch denaturiert ist. Zudem liefert sie Informationen über den Einfluss intrinsischer und extrinsischer Faktoren auf die Proteinstabilität. Die DSC gilt als die beste und genaueste quantitative Analyse der thermischen Stabilität in der Charakterisierung biopharmazeutischer Proteine. Sie wird zur Vorhersage der Langzeitstabilität eingesetzt.[1,10-14] Die mittels DSC ermittelten TM-Werte werden häufig als Parameter herangezogen, um die Stabilität bei der Kandidatenauswahl (hinsichtlich Entwicklungsfähigkeit), beim Formulierungsscreening und bei der Verfahrensentwicklung zu klassifizieren. Stabilere Proteine weisen einen höheren TM-Wert auf. Auch die mittels DCS gemessenen Größen Enthalpie (∆H), Tonset, T1/2 und ∆Cp werden für die Stabilitätsklassifizierung, die Validierung von DSC-Daten, die quantitative Analyse der Proteinentfaltung und das HOS-Fingerprinting verwendet.[10-14]
DSC-Analysen von Proteinen unter definierten Lösungsbedingungen sind reproduzierbar und quantitativ, wenn die analysierten Proteine gleich oder sehr ähnlich sind (Abbildung 4). Das heißt, die DSC-Thermogramme weisen ähnliche Profile auf, und die Parameter (einschließlich TM, ∆H und Tonset) liegen innerhalb eines bestimmten Streubereichs.[12-14] Falls die Thermogramme unterschiedlich sind und sich die mittels DSC ermittelten Parameter ändern, weist dies auf Veränderungen der konformationellen Stabilität hin. Verursacht werden können diese durch Proteinfehlfaltung, Degradation, Aggregation, Lösungsmittelunterschiede, Änderungen der posttranslationalen Modifikation oder andere Änderungen der höheren Ordnungsstruktur.
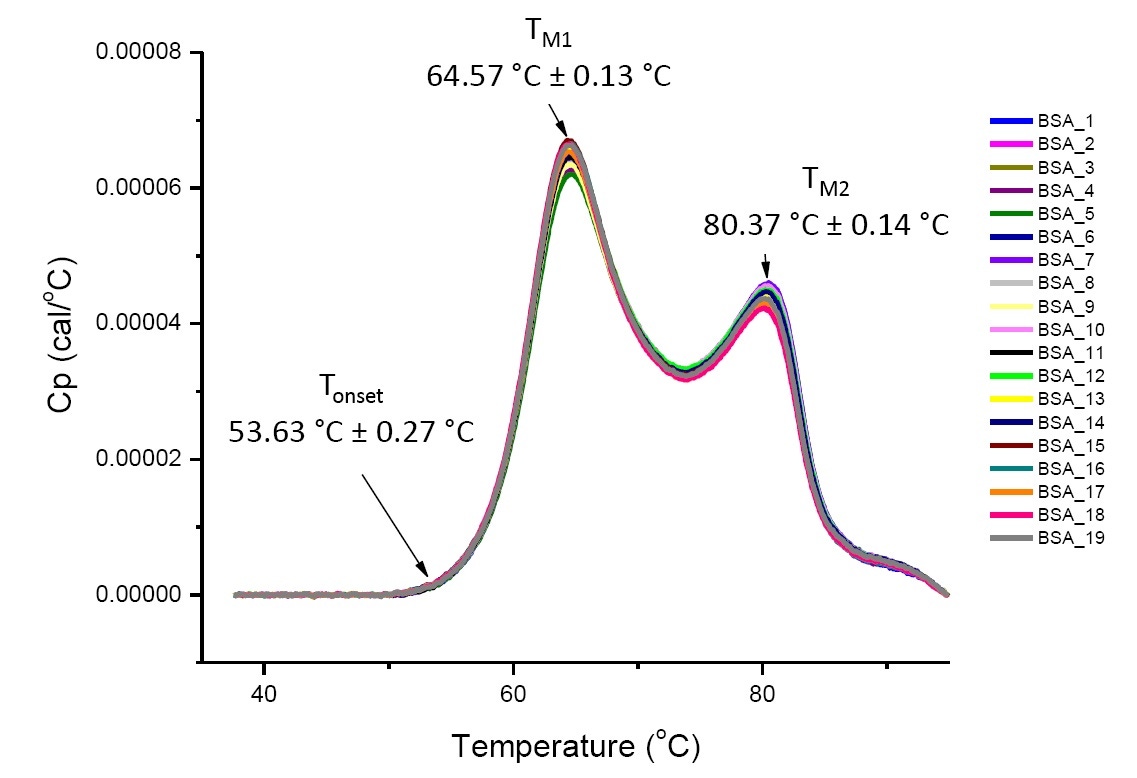
Abbildung 4. Neunzehn DSC-Thermogramme von Rinderserumalbumin (Sigma A1933, chromatographisch gereinigt) in PBS. Auftrag der DSC-Daten nach Normalisierung der Durchlaufrate, Puffer-Puffer-Subtraktion und Subtraktion der Integrationsbasislinie. Angegeben sind Mittelwert und Standardabweichung von Tonset, TM1 und TM2.
Aufgrund der reproduzierbaren und quantitativen Ergebnisse ist die DSC ein wertvolles HOS-Analysewerkzeug. Eingesetzt werden kann sie für die Produktevaluierung bei der Herstellung (einschließlich von Chargen- und Standortvergleichbarkeit), den Vergleich von Proteinvarianten und modifizierten Produkten (einschließlich von strukturellen Veränderungen durch Glykosylierung und Oxidation) und Biosimilaritätsstudien. Im Rahmen der HOS-Charakterisierung finden DSC-Daten auch Eingang in Dokumente im regulatorischen Umfeld im Rahmen der Zulassung neuer Wirkstoffe und Biosimilars. Bei einer Befragung von Forschern in der biopharmazeutischen Industrie stuften diese die DSC als „sehr hilfreiches“ bis „äußerst hilfreiches“ biophysikalisches HOS-Werkzeug bei Kandidatenauswahl, Formulierungsentwicklung, Produktcharakterisierung sowie Vergleichbarkeits- und Biosimilaritätsstudien ein.[15]
Die DSC-Thermogramme von Multidomänenproteinen, wie z. B. von Antikörpern, weisen mehrere Entfaltungsübergänge auf (Abbildung 2). Mit der DSC können die einzelnen Domänen charakterisiert und quantifiziert werden. Ebenso können die TM-Werte für zwei, drei oder mehr Übergänge bestimmt werden. Die TM-Werte entsprechen den Peaks im Thermogramm. Somit können sie einfach aus den DSC-Daten bestimmt werden, ohne dass eine komplexe Datenanalyse notwendig ist. Andere biophysikalische Assays zur Bestimmung der TM-Werte, wie z. B. CD, IF und DSF, können bei Multidomänenproteinen u. U. nur den ersten TM-Wert detektieren (der bei der niedrigsten Temperatur auftritt) oder den „dominantesten“ TM-Wert. Um aus spektroskopischen Daten oder Fluoreszenzdaten mehr als einen TM-Wert zu extrahieren; werden komplexe Datenanpassungen benötigt. Zudem ist diese Bestimmung u. U. nicht reproduzierbar.
Im Vergleich zu anderen Assays zur TM-Bestimmung wird für die DSC mehr Proteinprobenmaterial pro Durchlauf benötigt. Zudem kann der Durchsatz geringer ausfallen. Bei begrenzter Probenmaterialmenge kann daher zunächst eine TM-Klassifizierung mittels DSF oder IF durchgeführt werden. Anschließend können mehrere Proben ausgewählt werden, um die TM-Werte mittels DSC zu validieren. Bei Stabilitätsassays ist es wichtig, die TM-Ergebnisse mittels DSC zu validieren und sich bei der TM-Bestimmung nicht allein auf Fluoreszenz- oder Spektroskopieverfahren zu verlassen. Fluoreszenzbasierte Assays können Artefakte aufweisen. Diese beeinträchtigen das Ergebnis und können u. U. eine Verschiebung der TM-Werte zu höheren (oder niedrigeren) Temperaturen bewirken. Manche Proteine und Pufferbedingungen sind nicht kompatibel mit Fluoreszenzverfahren, sodass TM-Unterschiede durch diese Verfahren nicht detektiert werden können. Und schließlich können im Unterschied zur DSC mit Fluoreszenz- oder Spektroskopieverfahren nicht die kalorimetrische Enthalpie und andere thermodynamische Parameter bestimmt werden.
Die DSC wird in der biopharmazeutischen Industrie als maßgeblicher Thermostabilitätsassay erachtet, weil das Verfahren folgende Vorteile hat:
Beispiele zur Verwendung von DSC-Daten in der biopharmazeutischen Forschung und Entwicklung finden Sie in diesem Whitepaper, in anderen Whitepapers und Application Notes von Malvern Instruments und in den genannten Veröffentlichungen.
In der frühen Forschungsphase, in der es darum geht, ein Protein-Biopharmazeutikum zu identifizieren, liegt der Fokus zunächst auf der Entdeckung eines Zielmolekülsfür eine potenzielle Wirkstoffinteraktion. Sobald ein potenzielles Zielmolekül identifiziert wurde, konzentriert sich die Arbeit darauf, einen Wirkstoff zu finden, der mit diesem Zielmolekül interagiert. Dabei soll eine gewünschte biologische Wirkung erzeugt werden, die zu einem günstigen klinischen Ergebnis führt und einen Krankheitszustand verringert oder beseitigt. Nachdem ein oder mehrere Wirkstoffkandidaten identifiziert wurden, ist es wichtig, die Entwicklungsfähigkeit dieser möglichen biopharmazeutischen Kandidaten zu beurteilen.
Die Entwicklungsfähigkeit eines Proteinwirkstoffs umfasst eine Evaluierung der biophysikalischen Eigenschaften des Proteins. Hierdurch kann vorhergesagt werden, welche Proteine den Belastungen widerstehen, denen sie bei Zellkulturproduktion, Aufreinigung, Formulierung, Lagerung, Transport und Langzeitlagerung ausgesetzt sein können (Stabilität). Folgende Eigenschaften sind wichtig:
Es ist wichtig, die Analyse der Entwicklungsfähigkeit eines Proteinwirkstoffs nach Abschluss der Entdeckungsphase durchzuführen, bevor die präklinische Entwicklung beginnt. Sobald nämlich der Hauptkandidat ausgewählt ist und die Produktion hochgefahren wird, führen Änderungen des Wirkstoffs oder Verfahrens zu höheren Kosten und u. U. zur Einstellung des Projekts. Werden bereits in dieser frühen Phase gute Entscheidungen getroffen, hat dies für das Pharmaunternehmen weniger Ausfälle und eine erhebliche Kostenreduzierung zur Folge, und es können preisgünstige Arzneimittel produziert werden.
In dieser Phase der abgeschlossenen Entdeckung bzw. frühen Entwicklung sind für die biophysikalischen Assays oft nur begrenzte Mengen des gereinigten Proteins verfügbar. Auch ist die Zeit für die Assay-Evaluierung der Entwicklungsfähigkeit begrenzt. In dieser Phase werden alle gängigen biophysikalischen Verfahren verwendet, wie Fluoreszenz, Lichtstreuung, Größenausschlusschromatographie und DSC. Auch andere Assays, wie die analytische Ultrazentrifugation und der Wasserstoff-Deuterium-Austausch mit Massenspektrometrie (HDX-MS), können nützliche Informationen liefern.
Eine einfache frühe DSC-Analyse von Proteinwirkstoffen zur Klassifizierung der TM-Werte kann sehr hilfreich sein; um Wirkstoffkandidaten zu vergleichen, um Proteine mit optimaler Stabilität zu finden, um die Langzeitstabilität vorherzusagen und um Moleküle zu vermeiden, die thermisch weniger stabile Strukturen haben. Die Proteine mit den niedrigsten TM-Werten weisen eine größere Instabilität gegenüber Temperaturänderungen auf und sind daher als Wirkstoffkandidaten am wenigsten geeignet.
Doyle et al.[16] haben in einer Fallstudie biophysikalische Verfahren verwendet, um in der Entdeckungsphase und Kandidatenauswahl bispezifische Adnectin-Kandidaten zu charakterisieren. Zu den biophysikalischen Assays gehörten: SEC, AUC, SEC-MALS, Röntgenstrukturanalyse, SPR, DSF und DSC. Beim Vergleich zweier Adnectin-Kandidaten mittels DSC wies Kandidat B gegenüber Kandidat A einen höheren TM-Wert und einen höheren Tonset-Wert auf (was jeweils auf eine größere thermische Stabilität hinweist). Bei Kandidat B ergaben sich aus der DSC Hinweise auf eine Faltungsreversibilität: In der DSC-Messung wurde die Proteinprobe abgekühlt und wieder erwärmt. Dabei ergab der 2.Erwärmungszyklus ein übereinstimmendes DSC-Profil. Diese Reversibilität lässt auch auf verbesserte Expressionsniveaus und eine geringere Neigung zur Aggregatbildung schließen.
Die größte Proteinklasse bei auf dem Markt bzw. in klinischen Studien befindlichen Biopharmazeutika ist die der monoklonalen Antikörper. Monoklonale Antikörper zeigen in der DSC in der Regel ein komplexes Multidomänen-Thermogramm (Beispiel siehe Abbildung 2). Der größte, markanteste Peak entspricht der Fab-Domäne (dem antigenbindenden Fragment) des Antikörpers. Auch die CH2- und/oder CH3-Domänen können häufig beobachtet werden. Die relativen Positionen und TM-Werte der verschiedenen Domänen hängen von dem jeweiligen monoklonalen Antikörper ab. Sie können je nach Unterklasse und Optimierung variieren, wie die nachfolgenden Beispiele zeigen.
In einem 2007 veröffentlichten Artikel haben Garber und Demarest den Einsatz der DSC zur Charakterisierung von 17 vollständigen therapeutischen Antikörpern beschrieben.[17,18] Bei der Analyse mit dem MicroCal VP-Capillary DSC zeigten die Fab-Domänen dieser Antikörper thermische Entfaltungsübergänge mit Mittelpunkttemperaturen (TM) zwischen 57 °C und 82 °C. Die Autoren wiesen nach, dass IgG1 die stabilste humane Unterklasse war, gefolgt von IgG2 und IgG4. Anhand der verwendeten Beispiele zeigten sie, dass Stabilitätsunterschiede zwischen den Antikörpern einer ähnlichen Unterklasse auf die variablen Domänen zurückzuführen waren. Die spezifischen Merkmale der einzelnen V-Gene führten zu einem breiten Spektrum an Fab-Stabilitäten.
Manche Antikörper-Fab-Domänen können ausnehmend stabil sein, andere wiederum relativ instabil. Sehr niedrige Fab-Stabilitäten bzw. TM-Werte, wie sie bei mehreren Konstrukten in [17] beobachtet wurden, waren gegenüber den Antikörpern mit erhöhten Fab-TM-Werten mit einer schlechten/verringerten Antikörperexpression und erhöhten Aggregation verbunden. Infolgedessen ist es schwieriger, Proteine mit niedrigeren TM-Werten zu reinigen. Daher ist bei der Verfahrensentwicklung mit einer verminderten Ausbeute und einer erhöhten Wahrscheinlichkeit einer Aggregation bei Lagerung zu rechnen. Die Daten aus dieser Studie[17] belegten, dass DSC-Stabilitätsdaten ein nützliches Instrument zur Kandidatenauswahl für Biopharmazeutika darstellen.
Ionescu et al.[19] haben DSC-Profile von drei humanisierten monoklonalen IgG1-Antikörpern und deren Fab- und Fc-Fragmenten (nach enzymatischem Abbau) bei neutralem pH-Wert erstellt (mit dem MicroCal VP-Capillary DSC). Von einigen Ausnahmen abgesehen zeigten die Thermogramme, dass der Übergang mit dem größeren experimentellen Enthalpiewert auf den Beitrag der Fab-Fragmente zurückzuführen war (ähnlich zu den Ergebnissen aus [17]). Auch die TM-Werte variierten erheblich, selbst für Fab-Fragmente, die von derselben humanen Keimbahn stammten. Ionescu at al. schlugen vor, die Entfaltungsenthalpie als Schlüsselparameter für die Erkennung von Entfaltungsereignissen im Schmelzprofil eines intakten IgG1-Antikörpers zu verwenden. Einige DSC-Thermogramme wiesen zwei Übergänge auf. Der erste Übergang repräsentierte die Entfaltung des Fab-Fragments und der CH2-Domäne und der zweite Übergang die Entfaltung der CH3-Domäne. Bei anderen Antikörpern repräsentierte der erste DSC-Übergang die Entfaltung der CH2-Domäne und der zweite Übergang die Entfaltung des Fab-Fragments und der CH3-Domäne. Es gab andere Fälle, in denen das DSC-Profil drei Übergänge aufwies und die Fab-Entfaltung bei Temperaturen auftrat, die sich von den Schmelzübergängen der CH2- und CH3-Domänen unterschieden. Eine niedrige Stabilität oder Heterogenität des Fab-Fragments kann hinsichtlich der Langzeitlagerung und Produktionskonsistenz problematisch sein. Nach Meinung der Autoren ist ein umfassendes Verständnis der Merkmale eines DSC-Profils entscheidend für die Kandidatenauswahl in den frühen Phasen der Entwicklung therapeutischer monoklonaler Antikörper.
Die Arbeiten, die in den zwei oben genannten und weiteren, zu gleicher Zeit veröffentlichten Artikeln beschrieben sind, bildeten die Basis für nachfolgende Fallstudien und für den Einsatz der DSC und anderer Untersuchungen zur Stabilitätscharakterisierung bei der Auswahl biopharmazeutischer Kandidaten.
Um die thermische Stabilität von Wirkstoffkandidaten genauer zu bestimmen, werden Proteine häufig unter mehreren pH-Bedingungen analysiert, um eventuelle Unterschiede aufzudecken. Diese unterschiedlichen pH-Werte können Bedingungen nachbilden, wie sie in Formulierungen und/oder Prozesspuffern bei der Herstellung des Wirkstoffs vorliegen.
Jiang et al.[20] haben in einer Fallstudie den Einsatz einer ausgewählten HOS-Charakterisierung zur Evaluierung der Entwicklungsfähigkeit (Fähigkeit zur Herstellung sowie zur Aufrechterhaltung der allgemeinen Produktqualität) zweiter potenzieller monoklonaler Antikörperarzneimittel beschrieben. Bei den Antikörpern X und Y handelte es sich um monoklonale IgG3-Antikörper gegen das gleiche Zielmolekül, die ähnliche biologische Aktivitäten zeigten. Unter Einsatz einer Kombination von biophysikalischen Methoden (einschließlich DSC, Nah-UV-CD, FTIR, DLS, Fluoreszenzspektroskopie und SEC) untersuchten die Autoren:
Die sauren Bedingungen (pH 3) wurden gewählt, um die Puffer nachzubilden, die bei der Antikörperaufreinigung und der viralen Inaktivierung verwendet wurden. Mit dem pH-5-Puffer wurden die Lagerungsbedingungen (Formulierung) simuliert, und die pH-7-Puffer wurden während der gesamten Aufreinigung eingesetzt.
Messungen der beiden mAb-Kandidaten in pH-7-Puffer (PBS) mit dem MicroCal VP-Capillary DSC zeigten zwei Übergänge: Die CH2/Fab-Domänen entsprachen dem ersten Übergang, und die Entfaltung der CH3-Domäne entsprach dem zweiten Übergang.[20] mAb Y war bei ph 7 thermisch geringfügig stabiler. Dies konnte aus den höheren TM-Werten der DSC und der höheren Anfangstemperatur (Tonset) geschlossen werden. In pH-3-Puffer (C3N) wiesen beide Kandidaten in der DSC nur einen thermischen Übergang auf. Dies lässt darauf schließen, dass die Proben bei der Ausgangstemperatur infolge des niedrigen pH-Werts bereits partiell entfaltet waren. Wie erwartet waren die thermischen Übergangstemperaturen und Entfaltungsenthalpien beider Kandidaten bei pH 3 (C3N) deutlich geringer als bei pH 7 (PBS). In C3N wies jedoch mAb X (TM 63,2 °C) eine bessere thermische Stabilität auf als mAb Y (TM 48,2 °C), wenngleich bei neutralem pH-Wert mAb Y thermisch stabiler war. Obwohl dieses Ergebnis untypisch ist, steht es im Einklang mit den Nah-UV-CD-Daten. Diese zeigten, dass mAb X bei pH 3 mehr Tertiärstruktur beibehielt. Die durch pH 3 induzierten Veränderungen der thermischen Stabilität der beiden Kandidaten schienen nach Dialyse in PBS vollständig reversibel zu sein.
Insgesamt zeigten die DSC-Ergebnisse, dass beide Proteine bei neutralem pH-Wert sehr stabil waren und dass die thermische Stabilität von mAb Y höher war als die von mAb X in PBS, jedoch niedriger als die von mAb X in C3N.[20] Die CD- und DSC-Daten zeigten, dass mAb Y bei pH 3 größere strukturelle Veränderungen erfuhr als mAb X. Die durch pH 3 induzierten Veränderungen der thermischen Stabilität waren bei beiden mAbs vollständig reversibel. Veränderungen der Tertiärstruktur bei mAb Y schienen jedoch zumindest teilweise irreversibel zu sein. Gegenüber mAb X zeigte mAb Y eine bessere thermische Stabilität bei neutralem pH-Wert und eine verbesserte Lagerstabilität in seinem Formulierungspuffer bei pH 5 und 37 °C. Zusammenfassend wiesen die Ergebnisse darauf hin, dass für die Prozessstabilität (Robustheit gegenüber pH-Änderungen) und die Lagerstabilität (Einfluss erhöhter Temperaturen) unterschiedliche Determinanten vorliegen.
Da der gefaltete mAb Y in PBS bei pH 7 eine höhere thermische Stabilität und bei pH 5 und 37°°C eine bessere Lagerstabilität aufwies, wurde mAb Y für die Übernahme in die weitere Entwicklung vorgeschlagen.[20] Nach einer Inkubation bei niedrigen pH-Werten (in PBS) wurden ein Zusammenbruch der Tertiärstruktur und eine anschließende Selbstassoziation beobachtet. Beide Prozesse waren irreversibel. Daher wurde empfohlen, nicht nur mAb Y, sondern auch den Aufreinigungsprozess weiterzuentwickeln, um die Exposition von mAb Y gegenüber pH 3 zu minimieren. Diese Fallstudie veranschaulicht, wie Stabilitätsdaten im Rahmen der Kandidatenauswahl und Verfahrensentwicklung helfen, die durch das Verfahren erzeugte Menge an irreversiblen Aggregaten und konformationellen Varianten zu minimieren und letztendlich die Verfahrensausbeute zu erhöhen.[20]
Weitere Fallstudien zur Evaluierung und Vorhersage der Entwicklungsfähigkeit auf Basis der thermischen Stabilitätsdaten aus der DSC werden von Satish et al zusammengefasst.[21]
Tavakoli-Kenshe et al.[22] wollten herausfinden, anhand welcher anderen Prädiktoren, abgesehen von Stabilitätsuntersuchungen, die Entwicklungsfähigkeit vorhergesagt werden kann. Sie untersuchten fünf Varianten von IgG1- und IgG4-Antikörpern. Mit einem Plattenrheometer erzeugten sie definierte Scherbedingungen bei einer bekannten Luft-Flüssigkeitsgrenzfläche, um die Stabilität in dieser Umgebung zu messen. Anhand der Daten des Rheometers wurden die Antikörper nach Stabilität klassifiziert. Diese Ergebnisse wurden mit beschleunigten thermischen Stabilitätsdaten und der mit dem MicroCal VP-Capillary DSC gemessenen Schmelztemperatur der CH2-Domäne (TM1) verglichen, um die Komplementarität der Verfahren zu untersuchen. Die Ergebnisse wiesen darauf hin, dass die Verfahren orthogonal sind. Die thermischen Verfahren basieren auf intermolekularen Wechselwirkungen. Die mittels Rheometer gemessene Stabilität basieren auf lokalen Entfaltungen, durch die die weniger stabilen, die Aggregation begünstigenden Bereiche aufgedeckt werden. Durch molekulare Modellierung können die Auswirkungen der Veränderungen auf die Antikörperstrukturen gezeigt werden. Es zeigt sich ein möglicher Beitrag der Fc-Konformation und der Fab-Fc-Kopplung bei der Bestimmung der Stabilität der suspendierten Proteine. Die Daten belegen, dass Scherversuche als potenzieller orthogonaler Stabilitätsindikator dienen können, komplementär zu herkömmlichen thermischen Verfahren wie der DSC. Gemeinsam ermöglichen sie die Auswahl von Proteinkandidaten mit verbesserter Stabilität für den Aufreinigungsprozess.
In der biopharmazeutischen Forschung werden die Treffermoleküle oder parentalen Moleküle eines Proteinwirkstoffs häufig modifiziert bzw. optimiert, um ihre biophysikalischen Eigenschaften und die Entwicklungsfähigkeit des Proteins zu verbessern. Nach erfolgter Optimierung ist es wichtig, noch vor der Kandidatenauswahl die Stabilität der verschiedenen optimierten Proteine zu evaluieren, um zu überprüfen, ob die Optimierung zu negativen Auswirkungen geführt hat. Ein Beispiel dafür wurde in einer Malvern Instruments Application Note[23] gezeigt. Ein MicroCal VP-Capillary DSC diente als Screening-Werkzeug für den parentalen Antikörper und zwei optimierte Antikörper, um die thermische Stabilität und die Entwicklungsfähigkeit der optimierten Konstrukte besser vorhersagen zu können. Auch die Aggregationsbildung nach beschleunigter Lagerung wurde untersucht (mittels SEC). Bei einer beschleunigten Stabilitätsuntersuchung zur Unterstützung der Kandidatenauswahl korrelierte der optimierte Antikörper mit dem am stärksten herabgesetzten TM-Wert mit dem Antikörperkonstrukt mit dem höchsten Aggregationsgrad.
Demarest et al. haben ein instabiles Fab-Fragment optimiert und mit einem MicroCal VP-Capillary DSC die verbesserte Proteinstabilität demonstriert.[18, 24] In dem Bestreben, Stabilisierungsstrategien für Antikörper und Antikörperfragmente zu entwickeln, wählten sie ein Fab-Fragment mit ungünstigem Verhalten aus. Dieses erkannte das Tetanustoxin (αTT) und wurde ursprünglich aus Humanmaterial isoliert. Anschließend wandten sie eine Mutagenesestrategie an, um dem Fab-Fragment wieder Stabilität zu verleihen. Die Autoren wählten zwecks Randomisierung 45 Restpositionen aus. Fast 4500 einzelne Kolonien wurden entnommen und in Expressionsmedium kultiviert. Die die exprimierten Fab-Fragmente enthaltenden Überstände wurden bei drei erhöhten Temperaturen (70 °C, 72 °C und 74 °C) Hitzestress ausgesetzt. Varianten, die eine erhöhte Thermostabilität zeigen, wurden einer erneuten Thermotoleranzuntersuchung unterzogen, um ihre Eigenschaften zu bestätigen. (Eine vollständige Liste der stabilisierenden Mutationen ist in [24] enthalten.) Ca. 1 % der Varianten in der Bibliothek zeigte eine erhöhte Thermostabilität. Von den Treffern befanden sich 14 in der VH-Domäne und die verbleibenden vier in der VL-Domäne. Dieses Ergebnis legte nahe, dass die Stabilität des nativen Fab-Fragments durch die marginale Stabilität der VH-Domäne begrenzt wurde. Erstaunlicherweise wurde in der ca. 2000 Mitglieder umfassenden Bibliothek der konstanten Domänen kein Mutant gefunden, um das Fab-Fragment als Ganzes zu stabilisieren. Die Autoren vermuteten, dass innerhalb der konstanten Domänen tatsächlich stabilisierende Mutationen vorliegen, dass jedoch die begrenzte Stabilität der VH-Domäne die Temperatur bestimmt, bei der sich das Fab-Fragment entfaltet, und es daher nicht möglich war, solche Ereignisse zu beobachten.
Innerhalb der αTT-VL-Domäne wurden vier stabilisierende Mutanten entdeckt.[24] Die Mutation des VK4-Konsensusresiduums W50 zu Ala (dem Konsensusresiduum für VK1) oder His war hochgradig stabilisierend. Histidin kommt an Position 50 in humanen VK-Domänen selten vor, häufig jedoch in humanen VL-Domänen. Dieses Residuum befindet sich in der Nähe der VH/VL-Domänengrenzfläche. Die Autoren nahmen an, dass sein Beitrag zur Fab-Stabilität mit einer Verstärkung der VH-Domäne zusammenhängen könnte, da insbesondere die VH-Domäne die Stabilität des αTT-Fab-Fragments zu begrenzen schien. Untersuchungen der isolierten VL-Domäne zeigten jedoch abweichende Ergebnisse.
Es wurden 12 Konstrukte generiert, die zwischen 3 und 11 der im ersten Screening identifizierten stabilisierenden Mutationen enthielten.[24] Verschiedene Kombinationen wurden rational abgeleitet, um den apparenten Beitrag der einzelnen Mutanten zur Fab-Stabilisierung zu bestimmen. In einem parallelen Transformations-/Expressionsexperiment wurde durch die Einführung mehrerer stabilisierender Mutationen in das αTT-Fab-Fragment die Fab-Expression verstärkt. Dies lässt darauf schließen, dass die stabileren Konstrukte eine vermehrte Expression in den Zellen zur Folge haben. Die besten Konstrukte zeigten konsistent mehr als dreifache Erhöhungen des Expressionsertrags gegenüber dem Wildtyp. Die Stabilität der einzelnen Fab-Fragmente wurde mittels DSC und CD-Spektroskopie (CD) evaluiert. Anhand der gemessenen Tm-Werte wurde die apparente Fab-Stabilität klassifiziert.
Ein wichtiger Aspekt war die potenzielle Auswirkung dieser Mutationen auf die Antigenbindungsfunktion des αTT-Fab-Fragments. Die thermostabilisierenden Mutationen wurden aus dem ursprünglichen Screening abgeleitet. Mittels quantitativem ELISA wurde sowohl die CL-Domäne als auch der Histidin-Tag am C-Terminus von CH1 entdeckt. Die Autoren stellten fest, dass die thermostabilisierenden Mutationen die apparente Affinität des αTT-Fab-Fragments in einem funktionellen ELISA gegenüber dem Wildtyp erhöhten.[24] Die funktionelle Kapazität der einzelnen Fab-Varianten schien im direkten Zusammenhang mit ihrer Stabilität zu stehen, ausgedrückt durch den Tm-Wert aus der DSC. Dass die funktionelle Aktivität und die Proteinexpression mit den TM-Werten aus der DSC korreliert werden konnten, lässt darauf schließen, dass die Auswahl des stabilsten Konstrukts zu einer verbesserten Entwicklungsfähigkeit führt.
Seeliger et al.[25] haben einen Antikörper, der in vitro eine Aggregationsneigung zeigte, mithilfe computerbasierter Designstrategien systematisch modifiziert. Die daraus entstandene Reihe eng verwandter Antikörper, die aus einem Wildtyp-Antikörper mAb1 optimiert wurden, zeigten in der Beurteilung mit biophysikalischen Verfahren (u. a. mit dem MicroCal VP-Capillary DSC) und in Langzeitstabilitätsversuchen eine verbesserte Stabilität. Wie Demarest et al. bei dem oben beschriebenen Beispiel mit dem αTT-Fab-Fragment[24] feststellten, erhöhten sich die Expressionsniveaus der mutierten Proteine auch gegenüber dem Wildtyp-Kandidaten. Zusammengefasst zeigen die experimentellen und rechnerischen Daten aus dieser Studie, wie computerbasierte Verfahren die Antikörperoptimierung hinsichtlich einer höheren Stabilität unterstützen können.
Die DSC-Analyse des ursprünglichen Wildtypproteins ergab einen TM-Wert von 68 °C für den ersten Übergang des Thermogramms (der der Fab-Domäne zugeordnet werden konnte). In den DSC-Thermogrammen der schwerkettigen oder leichtkettigen Mutationen stieg der TM-Wert des ersten thermischen Übergangs von 68,9 °C auf 72,8 °C an. Bei dem Mutanten mit optimierten schweren und leichten Ketten wurde das erste Endotherm bei 70,5 °C beobachtet, mit dem größeren Peak (Fab) bei 83,5 °C. Der TM-Wert für die Entfaltung des Antikörper-Fab-Fragments erhöhte sich nach Entfernung der vorhergesagten entfaltungsspezifischen Eigenschaften in den schweren und leichten Ketten um 16 K. Wurde die Entfaltung anhand der Veränderung der Tryptophanfluoreszenz überwacht, konnte derselbe Trend beobachtet werden.[25]
Die Computersimulation sagte voraus, dass die meisten eingeführten Mutationen die thermodynamische Stabilität erhöhen würden. Diese Ergebnisse standen im Einklang mit denen aus den DSC- und RALS-Messungen (Rechtwinkellichtstreuung). Beide Versuche zeigten eine kontinuierliche Zunahme der Stabilität mit zunehmender Anzahl von Mutationen. In beiden Versuchen stellten die Autoren zudem fest, dass die Optimierung der leichten Kette eine merkliche Auswirkung auf die Stabilität hatte. Diese Feststellung steht im Einklang mit den berechneten Veränderungen der Thermostabilität für die einzelnen Mutationen.[25]
Alle optimierten Varianten von mAb1 wurden mit Titern über 50 mg/l exprimiert, während die mAb1-Titer beim Wildtyp unter 1 mg/l lagen. Der dramatische Anstieg der Expressionsniveaus bei den Varianten, die nur eine optimierte Kette enthielten, war aus den DSC-Ergebnissen nicht vorhersagbar, die bei diesen Varianten eine geringe Stabilitätszunahme zeigten. Nach den Berechnungen sollte die Stabilität der einzelnen Immunoglobulindomänen mit den eingeführten Mutationen jedoch verbessert werden. Aus Erhitzungsversuchen erscheint es plausibel, dass die Stabilität jeder einzelnen Kette der limitierende Faktor ist. Demnach wären die beobachteten Effekte kinetisch und keine thermodynamischen Gleichgewichtseffekte. Sobald sich eine Domäne entfaltet, verschwindet der stabilisierende Effekt der Komplexbildung. Infolgedessen beginnt auch die zweite Kette, sich zu entfalten. Für die Proteinproduktion und die Entfaltung in vivo könnte die Existenz einer stabil gefalteten Domäne, die als Keimpunkt für eine weniger stabile Kette dient, ausreichend sein, um die Menge an erfolgreich gefalteten Antikörpern deutlich zu erhöhen.[25]
Für Unternehmen, die biopharmazeutische Arzneimittel entwickeln, ist es interessant, ob die schrittweise erhöhte Proteinstabilität (wie mit DSC und anderen biophysikalischen Assays gemessen) auch eine schrittweise Verbesserung der Langzeitstabilität und Haltbarkeit ergibt. Die Autoren führten mit den optimierten Varianten eine beschleunigte Stabilitätsuntersuchung bei 40 °C durch. Dabei stellten sie anhand des mittels SEC bestimmten Monomergehalts wesentliche Verbesserungen der Langzeitstabilität fest. Die vollständig optimierte Variante war zugleich die stabilste über den gesamten Versuchszeitraum von 4 Monaten.[23] Die thermodynamische Stabilität der variablen Domänen ist sicher ein wichtiger Faktor für die Langzeitstabilität. Doch anhand der Daten der Autoren und anderer veröffentlichter Daten zeigt sich auch, dass die thermodynamische Stabilität und die Haltbarkeit nicht in jedem Fall direkt korreliert werden können. Gegebenenfalls sind Anpassungen zur Verbesserung der Stabilität in der Formulierungsphase erforderlich.[25,26,27,28]
In neueren Veröffentlichungen wird der Einsatz von rationalem Design und der DSC und anderer biophysikalischer Werkzeuge zur Charakterisierung der Proteinstabilität beschrieben:
NISTmAb, ein Referenzmaterial eines monoklonalen Antikörpers des National Institute of Standards and Technology[35], wurde hinsichtlich der Entwicklungsfähigkeit evaluiert. Hierzu wurden biophysikalische HOS-Werkzeuge verwendet, wie sie typischerweise für mAb-Wirkstoffkandidaten zum Einsatz kommen. NISTmAb ist nicht für den Einsatz als therapeutisches Molekül bestimmt. Die Beurteilung der Entwicklungsfähigkeit von NISTmAb wurde repräsentativ für einen klassenspezifischen IgG1-mAb durchgeführt.[36] NISTmAb wurde unter unterschiedlichen Pufferbedingungen hinsichtlich Stabilität und Integrität evaluiert. Dabei wurden die Verfahren Elektrophorese, SEC-MALS, DLS und DSC eingesetzt. Die thermische Stabilität der NISTmAb-Probe in PBS-Puffer wurde mit dem MicroCal VP-Capillary DSC bestimmt. Die NIST-Probe zeigt drei thermische Hauptübergänge. Diese können der CH2-Domäne bei 71,2 °C, der CH3-Domäne bei 84,1 °C und der Fab-Domäne bei 88,9 °C zugeordnet werden.
Gegenüber den DSC-Profilen von IgG1-Molekülen aus [17] ist das NIST-Fab-Fragment stabiler (88,9 °C). Daher wurde der Schluss gezogen, dass das Molekül ein günstiges HOS-Profil aufweist.[36] Nach den vollständigen Daten der biophysikalischen Analyse weist NISTmAb günstige Voraussetzungen für die Entwicklung auf. Die beobachteten Veränderungen im Molekül nach Stressbedingungen stellen keine Risiken dar, die nicht mit der richtigen Formulierung und Herstellung in späteren Phasen des Entwicklungsprozesses gemindert werden können.
Die in diesem Whitepaper vorgestellten Ergebnisse zeigen deutlich die Bedeutung und Effizienz der DSC als biophysikalischer Stabilitätsassay bei der Auswahl biopharmazeutischer Kandidaten. Anhand der Ergebnisse der DSC und anderer Stabilitätsassays können biopharmazeutische Unternehmen fundierte Entscheidungen hinsichtlich der stabilsten und entwicklungsfähigsten Wirkstoffkandidaten treffen. Diese Kandidaten zeigen bei der Produktion und Aufreinigung die besten Resultate und weisen in der endgültigen Formulierung und als Arzneimittel weniger Probleme mit der Langzeitstabilität und Aggregation auf. Daraus ergeben sich eine kostengünstigere Arzneimittelproduktion und eine erhöhte Wahrscheinlichkeit, dass die endgültige Arzneimittelformulierung aktiv und stabil bleibt und ihre korrekt gefaltete Konformation beibehält.
Analytical Techniques for Biopharmaceutical Development, R. Rodriguez-Diaz, T. Wehr, S. Tuck (Hrsg.), Taylor & Francis, New York, USA (2005).
Biophysical Characterization of Proteins in Developing Biopharmaceuticals, D.J. Houde, S.A. Berkowitz (Hrsg.), Elsevier, Amsterdam, Niederlande (2015).
Biophysical Methods for Biotherapeutics: Discovery and Development Applications, T.K. Das (Hrsg.) John Wiley & Sons, Hoboken, NJ, USA (2014).
Biophysics for Therapeutic Protein Development, L.O. Nahri (Hrsg.), Springer, New York, USA (2013).
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 1. Monoclonal Antibody Therapeutics: Structure, Function, and Regulatory Space, J.E. Schiel, D.D. Davis, O.V. Borisov (Hrsg.), ACS Symposium Series Vol 1176 (2014). doi: 10.1021/bk-2014-1176.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 2. Biopharmaceutical Characterization: The NISTmAb Case Study, J.E. Schiel, D.D. Davis, O.V. Borisov (Hrsg.), ACS Symposium Series Vol 1201 (2015). doi: 10.1021/bk-2015-1201.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 3. Defining the Next Generation of Analytical and Biophysical Techniques, J.E. Schiel, D.D. Davis, O.V. Borisov (Hrsg.), ACS Symposium Series Vol 1202 (2015). doi: 10.1021/bk-2015-1202.