Die hier vorgestellte Arbeit veranschaulicht die Anwendung der dynamischen Differenzkalorimetrie (DSC) in den frühen Phasen der Proteincharakterisierung, um rasch wichtige Daten zur Proteinstabilität zu erhalten. Diese können als Richtwerte zur Unterstützung und Beschleunigung der Entwicklung von Flüssigformulierungen dienen.
Projekte zur Entwicklung monoklonaler Antikörper gibt es für eine große Vielzahl von klinischen Indikationen (1, 2). Rekombinante Proteine von kommerziellem Interesse, einschließlich monoklonaler Antikörper, müssen über ihre biologische Aktivität hinaus zusätzliche Eigenschaften aufweisen, um eine Entwicklung zu erfolgreichen Biopharmazeutika zu ermöglichen. Insbesondere müssen sie in einer gegebenen Umgebung gegenüber chemischer Degradation beständig und physikalisch stabil sein, ohne jegliche Tendenz zur Aggregation (3, 4). Des Weiteren müssen sie eine günstige Serumhalbwertszeit aufweisen und sollten kein oder ein sehr geringes immunogenes Potenzial zeigen (5).
Ein lyophilisiertes Arzneimittel ist nicht nur in der Herstellung teurer als Flüssigformulierungen, sondern muss vom Arzt auch rekonstituiert werden. Somit kann es 10 bis 20 Minuten dauern, bevor es dem Patienten parenteral verabreicht werden kann. Die vereinfachte Verabreichung ist der wichtigste Faktor, warum die biopharmazeutische Industrie Ressourcen auf die Entwicklung flüssiger Formulierungen konzentriert, anstatt auf herkömmliche lyophilisierte Arzneimittel. Bei den Flüssigformulierungen gilt es jedoch, einige technische Probleme zu überwinden. Die wichtigste Herausforderung besteht darin, das Protein-Biopharmazeutikum in einer Flüssigformulierung stabil zu halten, indem die physikalische Stabilität maximiert und die chemische Degradation minimiert wird. Besonders anspruchsvoll ist die Entwicklung von Biopharmazeutika für subkutane Verabreichung, weil hier die Flüssigformulierung eine möglichst hohe Proteinkonzentration aufweisen muss, um die aufgrund des möglichen Injektionsvolumens von 1,0 bis 1,5 ml bedingten Einschränkungen zu kompensieren (6).
|
Dies setzt die Formulierungsentwicklungsteams unter großen Druck, die fein abgestimmten Flüssigformulierungen zu generieren, die für das jeweilige Biopharmazeutikum optimal geeignet sind. Der Struktur- und Stabilitätsanalyse rekombinanter Proteine, insbesondere monoklonaler Antikörper, kommt erhebliche Bedeutung zu. Für die Entwicklung von Formulierungen für Biopharmazeutika ist sie eine unabdingbare Voraussetzung. Um strukturelle Informationen und Informationen zur Proteinstabilität zu erhalten, haben sich verschiedene Verfahren etabliert. Dabei hat sich die DSC zu einem der wichtigsten physikochemischen Verfahren zur Untersuchung der Stabilität von Protein-Biopharmazeutika entwickelt (7 bis 11). Die DSC ermöglicht die rasche Untersuchung der Proteinentfaltung ohne Markierung oder künstliche Sonden. Das Verfahren bestimmt die von einer Probe bei der Entfaltung eines Proteins aufgenommene Wärme, die ein Maß für dessen Thermostabilität und ein Kennzeichen für dessen Langzeitstabilität darstellt.
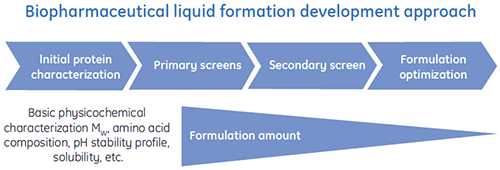
Es wurden spezielle Entwicklungsprogramme für Flüssigformulierungen konzipiert, um optimierte Flüssigformulierungen für Biopharmazeutika effizient zu generieren. Ein allgemeines Schema eines typischen Entwicklungsprozesses für Flüssigformulierungen ist in Abbildung 1 gezeigt.
Die DSC hat sich besonders in den frühen Entwicklungsphasen von Flüssigformulierungen bewährt. In diesen Phasen ist es wünschenswert, die Anzahl der Formulierungsansätze rasch zu verringern, um so Arzneimittelwirkstoffe und die für komplexe Analysen anfallende Zeit zu sparen.
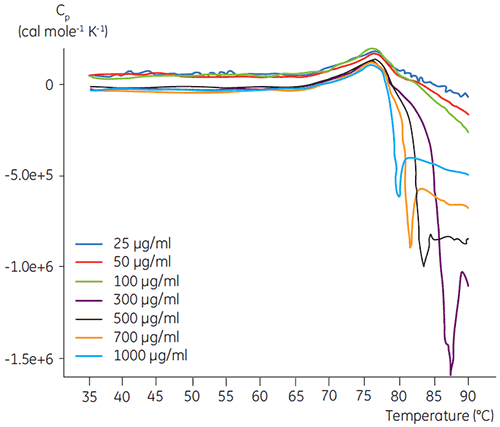
Die DSC-Experimente wurden unter Verwendung des Malvern MicroCal VP-DSC (Malvern Instruments) mit einer Durchlaufrate von 1,5 K/min durchgeführt. Vor der Analyse wurden die Proben fünf Minuten lang entgast. Bei allen Experimenten wurde die Referenzzelle des Kalorimeters mit einer dem Probenpuffer entsprechenden Pufferlösung befüllt. Aus den Proteinscans wurden die Pufferbasislinien subtrahiert. Für die Datenanalyse wurde die molare Wärmekapazität verwendet. Die temperaturinduzierte Entfaltung aller Proteine wurde auf Reversibilität überprüft, indem die erste DSC-Aufheizkurve mit der zweiten Aufheizkurve verglichen wurde (Daten nicht gezeigt). Es konnte keine Reversibilität festgestellt werden. Die Entfaltungstemperaturen wurden ermittelt, indem die kalorimetrischen Profile entsprechend einem Zwei-Zustands-Übergangsmodell analysiert wurden. Das optimale Signal-Rausch-Verhältnis wurde bei einer Antikörperkonzentration von 100 µg/ml erreicht (Abbildung 2). Wenngleich höhere Konzentrationen keine Auswirkungen auf die Entfaltungstemperatur hatten, induzierten sie eine starke Reaktionswärme infolge der Aggregation des Proteins nach der Entfaltung.
|
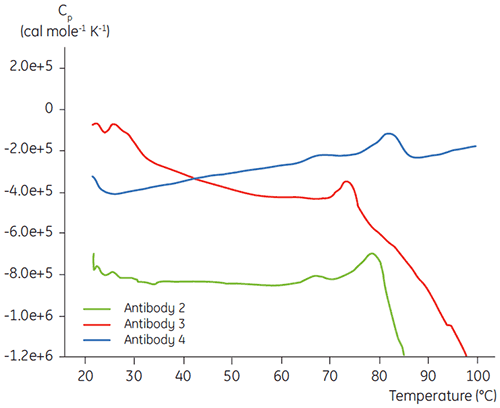
Innerhalb eines Entwicklungsprogramms für eine spezifische Antikörper Flüssigformulierung wird die DSC in der Anfangsphase der Proteincharakterisierung eingesetzt, um die Gesamtstabilität des Antikörpers mit den anderen, in Entwicklung befindlichen Antikörpern zu vergleichen. Wie aus Abbildung 3 ersichtlich, sind bei unterschiedlichen Antikörpern große Unterschiede bei den Entfaltungstemperaturen festzustellen.
|
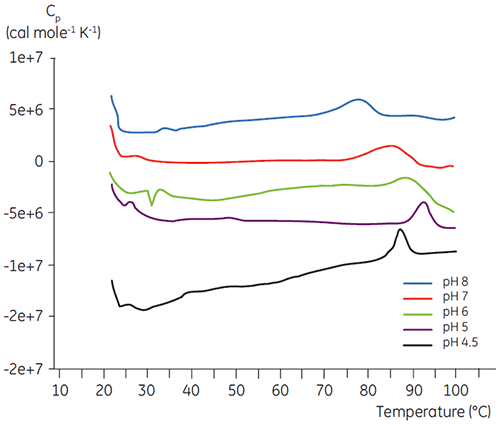
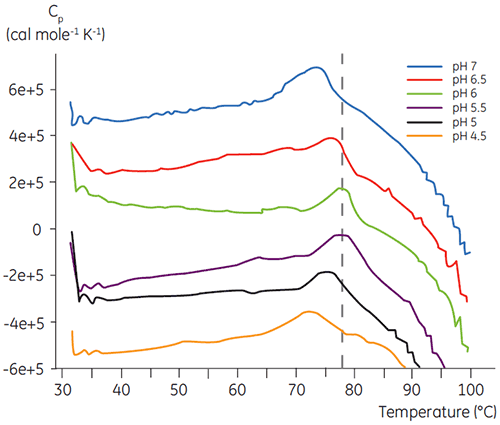
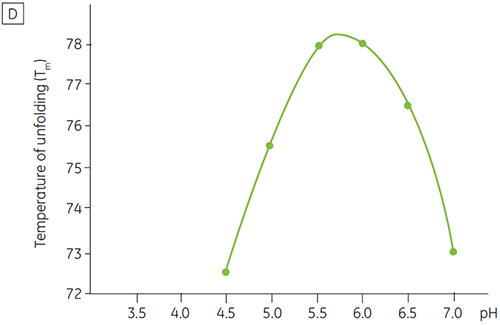
Wie die meisten Proteine sind Biopharmazeutika sehr pH-sensitiv. Für die Maximierung der Stabilität ist es daher entscheidend, den optimalen pH-Wert zu ermitteln. Die DSC hat sich als effizientes Instrument zur Optimierung des pH-Werts erwiesen: Es können Informationen gewonnen werden, ohne beschleunigte Stabilitätsuntersuchungen über zwei und vier Wochen durchzuführen. Die meisten Antikörper weisen die höchste Stabilität typischerweise zwischen pH 5,5 und pH 6 auf. Da jedoch Abweichungen festgestellt wurden, ist es dennoch wichtig, den optimalen pH-Wert zu charakterisieren. Die Abbildungen 4 und 5 zeigen die pH-Profile eines Fusionsproteins bzw. von Antikörper 2. Wie ersichtlich liegt der optimale pH-Wert für das Fusionsprotein (Abbildung 4) bei pH 5, mit einem resultierenden Tm-Wert von 92,8 °C. Das Fusionsprotein wurde bei pH-Werten über pH 5 folgerichtig destabilisiert. Es reagierte jedoch besonders empfindlich auf niedrige pH-Werte. Bei pH 4 konnte keine Entfaltung beobachtet werden (Daten nicht gezeigt). Abbildung 5 zeigt ein typisches pH-Profil eines Antikörpers. Die höchste Stabilität wurde bei pH 5,5 und pH 6 erzielt, mit einem resultierenden Tm-Wert von 78,5 °C.
|
|
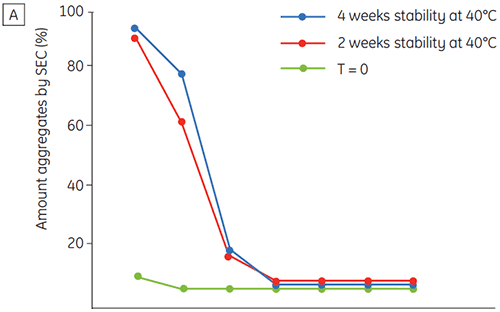
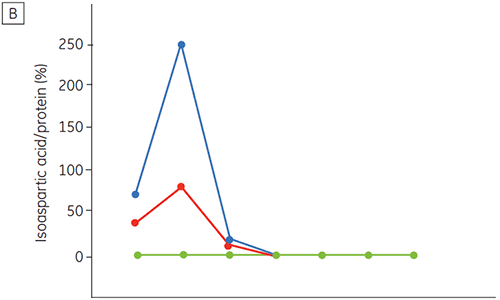
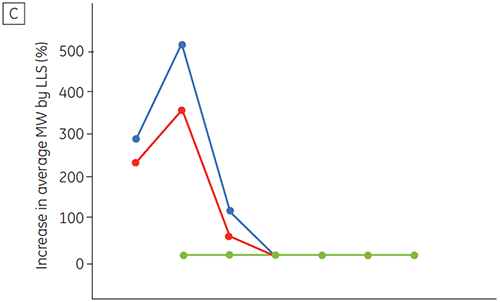
Abbildung 6 zeigt Daten zur chemischen und physikalischen Stabilität von Antikörper 2 als Funktion der Zeit und des pH-Werts (Anfangsphase der Proteincharakterisierung). Die Untersuchung des pH-Stabilitätsprofils zeigt, dass nach der Inkubation von Antikörper 2 über zwei und vier Wochen bei ≤ pH 5 und 40 °C deutliche Veränderungen der Stabilität eintreten. Mit der DSC konnte die optimale pH-Bedingung für diesen monoklonalen Antikörper ermittelt werden. Dabei wurden nur die frisch präparierten Proben verwendet (Nullzeit-Proben), und die erzielten Ergebnisse glichen denen der anderen Verfahren (Abbildung 5). Darüber hinaus konnten mit der DSC Werte größer gleich pH 6,5 als destabilisierend charakterisiert werden. Dies war innerhalb der zwei und vier Wochen dauernden Untersuchungen nicht erkennbar. Es hätte vielmehr einer acht und zwölf Wochen dauernden Stabilitätsuntersuchung bedurft, um diesen Effekt verdeutlichen. In anderen Worten: Die DSC-Ergebnisse entsprechen sehr gut den anderen Verfahren, die in dieser Untersuchung zur Optimierung des pH-Werts des Formulierungspuffers verwendet wurden.
|
|
|
|
Der Vorteil der DSC liegt darin, dass nur die Nullzeit-Proben analysiert werden müssen, um die Stabilität zu ermitteln. Dies beruht darauf, dass die die Erhitzung des Proteins bereits an sich stressinduzierend ist. Wenn die native Konformation eines Proteins stabilisiert wird, kann sie thermischen Belastungen üblicherweise in größerem Ausmaß widerstehen (höhere Entfaltungstemperatur) als ein Protein, das destabilisiert wird. Um lediglich Informationen über die Auswirkung einer spezifischen Flüssigformulierung auf die Proteinstabilität zu erhalten, war es somit bei Verwendung der DSC nicht erforderlich, die Proben auf Langzeitstabilität bei verschiedenen Temperaturen zu testen.
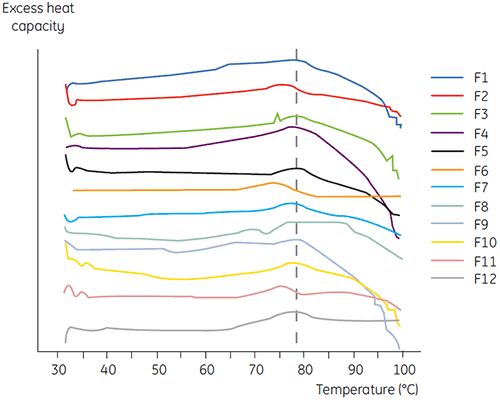
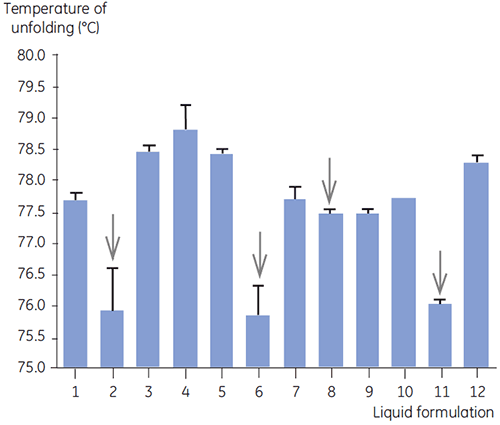
Die DSC wurde für das primäre Screening von Antikörper 2 eingesetzt, im Verbund mit anderen biophysikalischen Verfahren wie SEC, Laserlichtstreuung (LLS), Kapillarelektrophorese (CE) und SDSPAGE, um die vielversprechendsten Flüssigformulierungen auszuwählen. Die Proben wurden in eine beschleunigte Stabilitätsuntersuchung einbezogen und nach vier Wochen analysiert. Die DSC hat sich besonders zur Identifizierung der Formulierungen bewährt, die negative Auswirkungen auf Antikörper 2 hatten. Mit dem Verfahren konnten jedoch auch brauchbare Flüssigformulierungen identifiziert werden. Durch Analyse der Nullzeit-Proben identifizierte die DSC im primären Screening alle vier Flüssigformulierungen (Formulierung 2, 6, 8 und 11), die nach den Ergebnissen der anderen, in diesem Screening verwendeten Analyseverfahren als unzulänglich beurteilt wurden. Abbildung 7 zeigt die Thermogramme und Abbildung 8 die Entfaltungstemperaturen (Tm) von Antikörper 2 in den Flüssigformulierungen F1 bis F12 aus dem primären Screening. Die Entfaltung von Antikörper 2 in der Formulierung F9 ergab ein deutlich abweichendes Entfaltungsmuster. Sie wurde daher als kritische Formulierung beurteilt.
|
|
Die DSC ist ein Verfahren, das in den frühen Phasen der Proteincharakterisierung und Formulierungsentwicklung hilfreich eingesetzt werden kann. Es können rasch wichtige Daten zur Proteinstabilität gewonnen werden, die als Richtwerte zur Unterstützung und Beschleunigung der Entwicklung von Flüssigformulierungen dienen.
Autor dieser Application Note ist Dr. Fredrik Ollila, Novartis Pharma AG.