A estabilidade de uma proteína biofarmacêutica é fundamental para o sucesso ou a falha do desenvolvimento de um medicamento viável. A estabilidade da proteína afeta sua produção, fabricação, formulação, armazenamento a longo prazo, administração ao paciente e eficácia. As proteínas altamente estáveis são mais propensas a terem menos problemas durante o processo de fabricação, o custo de sua produção é mais econômico, e são mais prováveis a permanecem funcionais durante formulação e o armazenamento sem a alteração química ou agregação. Na abordagem QbD (Quality by Design) para o desenvolvimento de produtos biofarmacêuticos, a caracterização da estabilidade faz parte da avaliação inicial da “capacidade de desenvolvimento” e “viabilidade” de uma molécula candidata, bem como durante o desenvolvimento do processo e a fabricação. Os dados de estabilidade também estão incluídos na caracterização da estrutura de ordem superior (HOS) e nas "impressões digitais" utilizadas no apoio à produção, na comparabilidade biofarmacêutica e na avaliação de biossimilaridade. Além disso, a caracterização HOS de proteínas é "esperada" em envios regulatórios para novos medicamentos biofarmacêuticos e biologicamente similares.
Devido à natureza complexa das proteínas, as ferramentas de biofísica são importantes para a caracterização completa de um produto biofarmacêutico. Existe uma variedade de ferramentas biofísicas usadas para avaliar a estabilidade das proteínas, incluindo, entre outras, dicroismo circular (CD), espalhamento de luz dinâmico e estático (DLS e SLS), cromatografia por exclusão de tamanho com detector de espalhamento de luz multiangulo (SEC-MALS), espectroscopia no infravermelho por transformada de Fourier (FTIR), ultrafiltração analítica (ASC), cromatografia por exclusão de tamanho (SEC), fluorescência de varredura diferencial (DSF), fluorescência intrínseca (IF) e calorimetria de varredura diferencial (DSC).
Embora cada um desses ensaios biofísicos desempenhem um papel importante no desenvolvimento biofarmacêutico, a caracterização da estabilidade térmica por DSC é fundamental. Em um artigo de 2015 sobre técnicas biofísicas usadas para a caracterização da estrutura de ordem superior, Gokarn et al. afirmou que: “a DSC continua sendo uma técnica incomparável para avaliar a estabilidade termodinâmica de proteínas em uma determinada condição de tampão”[1].
A intenção deste documento é explicar como DSC é usada para caracterizar a estabilidade térmica de proteínas biofarmacêuticas (principalmente anticorpos) durante a seleção de medicamentos candidatos para avançar o seu desenvolvimento.
A DSC é uma técnica de microcalorimetria utilizada para caracterizar a estabilidade conformacional e térmica das proteínas, ácidos nucleicos, lipídios e outros biopolímeros [2-7]. A DSC mede a capacidade de aquecimento em função da temperatura. Os instrumentos DSC utilizados na caracterização de proteínas que estão descritos neste documento são instrumentos de “compensação de alimentação”, com o biopolímero na solução colocado em um fixo célula de amostragem e tampão correspondente colocado dentro de uma correspondência célula de referência. O sinal da capacidade de aquecimento (Cp) da célula de amostra é comparado com o da célula de referência. Conforme a temperatura das células aumenta, as diferenças de temperatura entre as células de referência e de amostra são continuamente medidas e calibradas a unidades de energia. A DSC é definida como um ensaio de “degradação forçada”; conforme a proteína é exposta ao aumento da temperatura, ela começa a se desdobrar e o Cp da proteína aumenta (Figura 1).
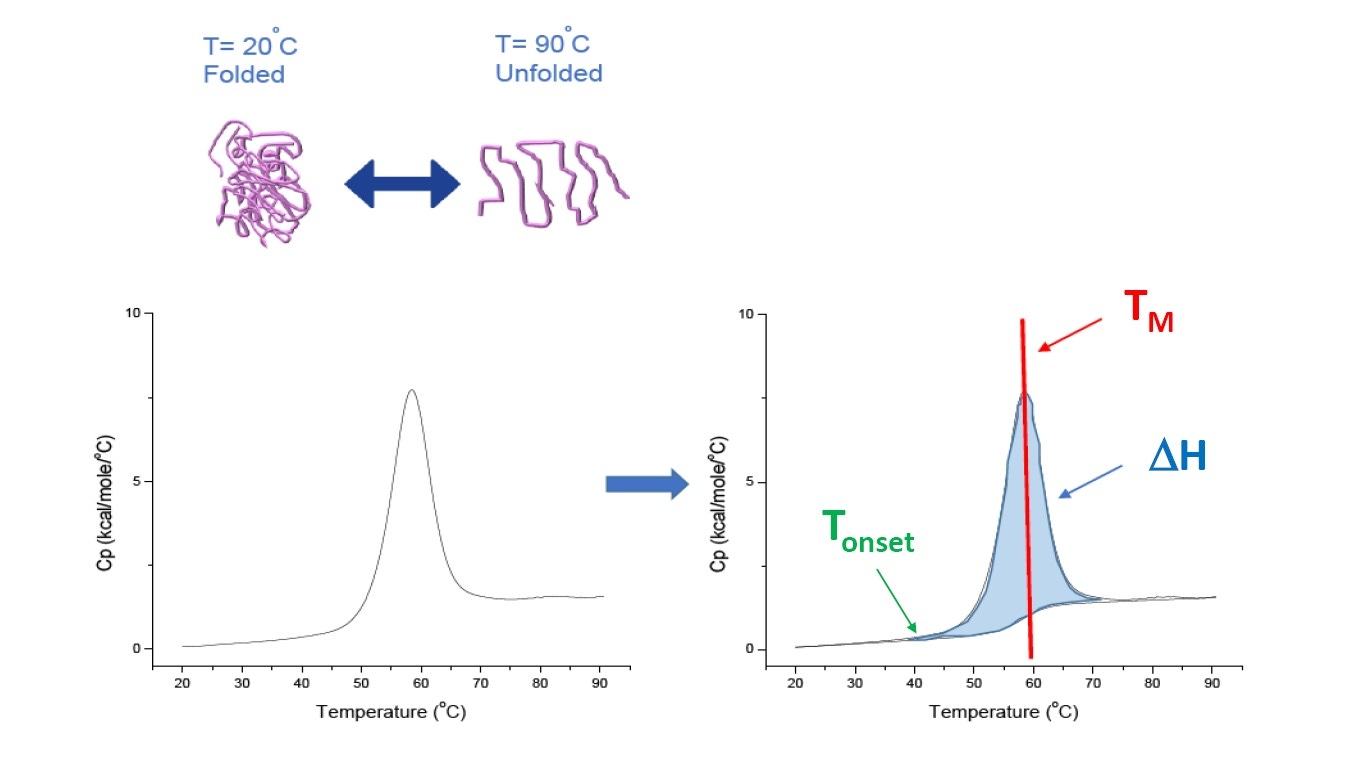
Figura 1: Como a DSC funciona. A capacidade de aquecimento (Cp) muda conforme a proteína desnatura termicamente. O experimento da DSC começa a uma temperatura na qual a proteína é primeiramente dobrada em sua conformação nativa. Com o aumento da temperatura, em algum momento a proteína vai começar a desdobrar/desnaturar (Tinício) e a Cp vai aumentar. Na temperatura em que 50% das proteínas estão em sua conformação nativa e 50% estão desnaturadas, a Cp atinge o seu valor máximo - este é o ponto médio de transição térmica, ou TM. Acima do TM, as proteínas estarão principalmente desnaturadas e, no fim do experimento de DSC, todas as proteínas estarão em sua conformação desdobrada. Os parâmetros experimentais para a DSC incluem o Tinício, o TM e a entalpia de desdobramento (ΔH).
A DSC mede diretamente a alteração da capacidade de aquecimento, sem a necessidade de fluorescência ou qualquer outra marcação ou sonda. O ponto médio de transição térmica (TM), também chamado de temperatura de fusão ou desnaturação, é a temperatura na qual 50% da proteína está em sua conformação nativa (dobrada) e 50% em sua conformação desnaturada. O TM é visto como o “pico” de um termograma de DSC. (Figura 1).
O TM é considerado um bom indicador da estabilidade térmica: quanto maior for o TM, mais termicamente estável estará a proteína. As proteínas de vários domínios (como os anticorpos) geralmente têm mais de um pico em um termograma de DSC; portanto, mais de um TM pode ser determinado (consulte a Figura 2 para obter um exemplo).
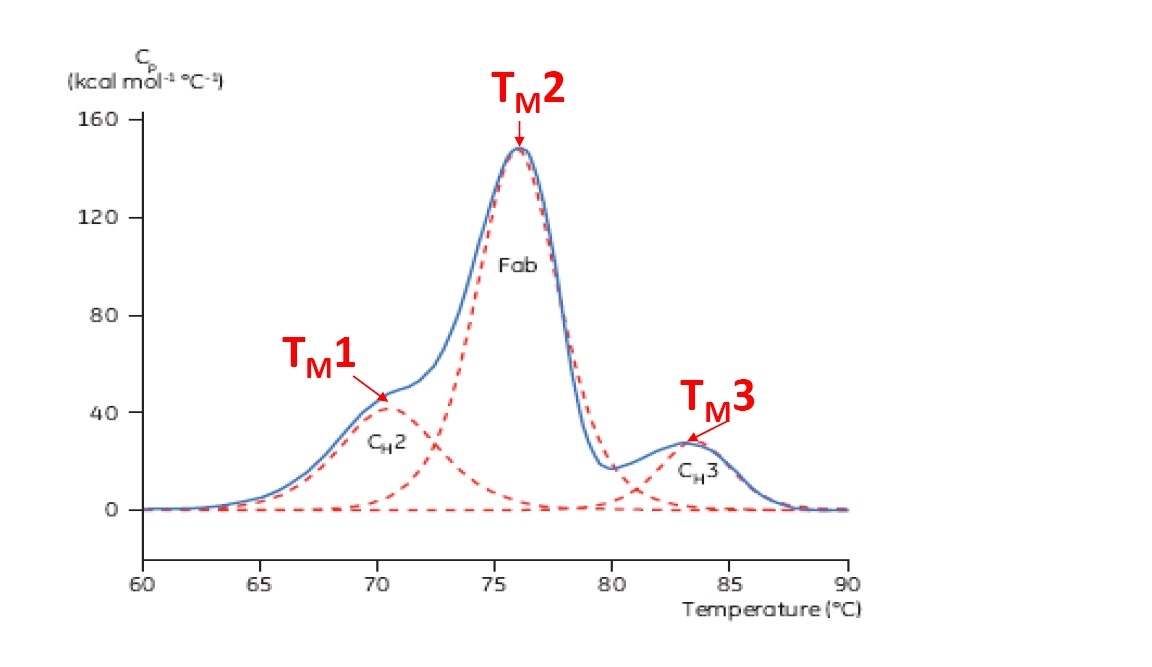
Figura 2: Termograma de DSC representativo de um anticorpo monoclonal, com os domínios CH2, Fab e CH3 identificados. As linhas vermelhas tracejadas são os picos com deconvolução da transição de cada domínio, com os três TMs indicados.
A DSC fornece outros parâmetros úteis, que podem ser usados para caracterizar e classificar a estabilidade das proteínas, incluindo a entalpia do desdobramento (ΔH), que é medida pelo área sob a curva. O desdobramento das proteínas é endotérmico, pois é necessária a aplicação de energia para quebrar as ligações não-covalentes secundárias que mantêm a proteína dobrada corretamente. A DSC também determina o Tinício (início do desdobramento), ΔCp (alteração da capacidade de aquecimento do desdobramento) e T1/2(largura à meia altura do pico, indicativo da forma do termograma do desdobramento). A análise por DSC pode incluir a determinação de qualquer combinação destes parâmetros.
A Malvern Instruments oferece o sistema de MicroCal VP-Capillary DSC[8,9], que é um Calorímetro de Varredura Diferencial (DSC) automatizado projetado para avaliação do TM e caracterização termodinâmica de proteínas e biopolímeros em solução.
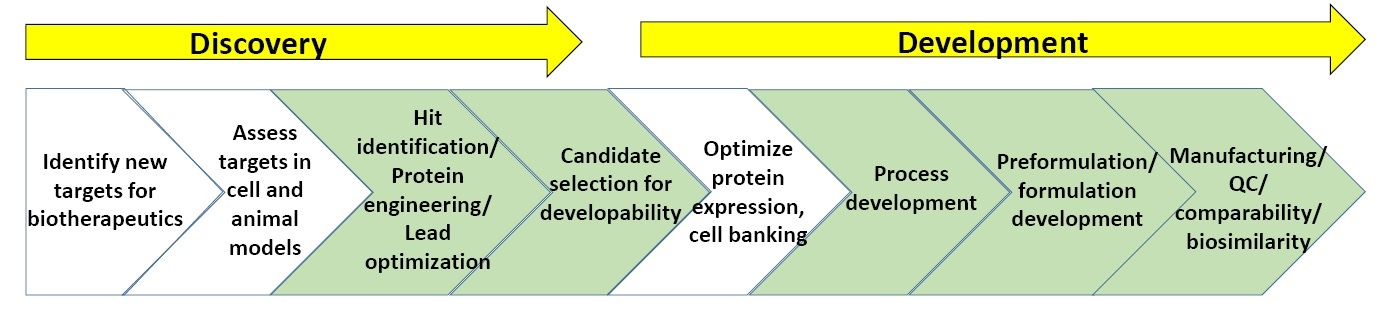
Figura 3: Esquema geral dos processos na descoberta e no desenvolvimento de produtos biofarmacêuticos.
A Figura 3 mostra um esquema geral dos processos de caracterização relacionados à descoberta e ao desenvolvimento de produtos biofarmacêuticos. As seções em verde mostram onde a caracterização biofísica, incluindo ensaios de estabilidade, normalmente é mais usada. No final deste documento encontra-se uma lista de "Leitura sugerida" na descoberta biofarmacêutica e desenvolvimento.
A sequência de aminoácidos, ou estrutura primária (1°), é o componente mais básico da corrente de polipeptídicas e da estrutura de proteínas. É importante compreender e caracterizar a estrutura tridimensional da proteína, também chamada de estrutura de ordem mais alta (HOS). Há três níveis de HOS de proteínas:secundário (2°), referindo-se aos locais padrões de dobragem da estrutura primária de uma proteína, incluindo α-helix, β-sheet, curvas e bobinas aleatórias,terciário (3°), a estrutura 3D de uma proteína que surge a partir seu leque de elementos estruturais secundários; e quaternário (4°), que descreve estruturas decorrentes da interação de dois ou mais idênticas ou diferentes cadeias polipeptídicas.
A fim de criar um desejável biofármaco, cientistas procuram inicialmente por biomoléculas que já demonstram uma elevada estabilidade durante a seleção dos candidatos, e, depois, podem inferir maior estabilidade através da engenharia das proteínas. Durante a purificação, normalmente ocorre a remoção das proteínas de condições em que estão estáveis, dobradas corretamente e ativas; portanto, é importante usar corretamente os tampões, aditivos, métodos de purificação e condições de armazenamento para manter a proteína o mais estável possível durante esse processo.
Quando as moléculas de proteínas são expostas a fatores estressantes, como calor, produtos químicos, alterações de pH, pressão, mistura e alta concentração, o que ocorre com frequência durante o processo de formulação biofarmacêutica e produção, suas conformações podem favorecer a forma desnaturada (desdobrada). Medicamentos a base de proteínas administrados por via subcutânea (SC) devem ser estáveis e permanecer inalterados em altas concentrações de proteínas (acima de 100 mg/mL) em seus recipientes, (por exemplo, frasco ou seringa pré-preenchida), geralmente por vários anos. As proteínas na solução também são suscetíveis a modificações como a desamidação e a oxidação, o que também pode levar a proteínas desnaturadas e inativas.
No caso de produtos biofarmacêuticos a base de proteínas, a desnaturação e outras modificações podem resultar na formação de agregados que levam a produtos com eficácia reduzida ou medicamentos não funcionais. Além disso, a agregação de proteína foi envolvida potencialmente em respostas imunogênicas possivelmente fatais em pacientes. Ao usar medicamentos com proteínas estáveis, obtém-se resultados de produção mais econômica e mais bem-sucedida, e produtos mais seguros e eficazes.
A DSC é uma medida da estabilidade conformacional de uma molécula e das alterações nas estruturas terciárias e quaternárias que ocorrem quando uma proteína é desnaturada termicamente, além de como os fatores intrínsecos e extrínsecos podem afetar a estabilidade das proteínas. A DSC é considerada a melhor e mais completa e análise quantitativa de estabilidade térmica usada na caracterização de proteínas biofarmacêuticas, como preditor de uma estabilidade de longo prazo[1,10-14] .. TMda DSC é um parâmetro que é frequentemente usado para ordem de classificação de estabilidade na seleção de candidatos (capacidade de desenvolvimento), análise de formulação e desenvolvimento do processo. Proteínas mais estáveis possuem maior TM. Entalpia (∆H), Tinício, T1/2 e ∆Cp da DSC também são usados ao classificar a estabilidade, a validação de dados DSC, a análise qualitativa do desdobramento da e a estrutura de ordem mais alta "impressões digitais' [10-14] ..
A análise de proteínas por DSC em condições de solução definidas é reproduzível e quantitativa se as proteínas analisadas forem iguais ou muito similares (Figura 4). Ou seja, os termogramas de DSC terão um padrão reproduzível e os parâmetros (incluindo TM, ΔH e Tinício) estarão dentro de uma faixa aceita[12-14]. Se os termogramas forem diferentes e os parâmetros de ajuste da DSC mudarem, isto pode sugerir que há dobramento incorreto da proteína, degradação, agregação, diferenças no solvente, alterações na modificação pós-translação ou outra diferença estrutural, causando alterações na estabilidade conformacional da proteína.
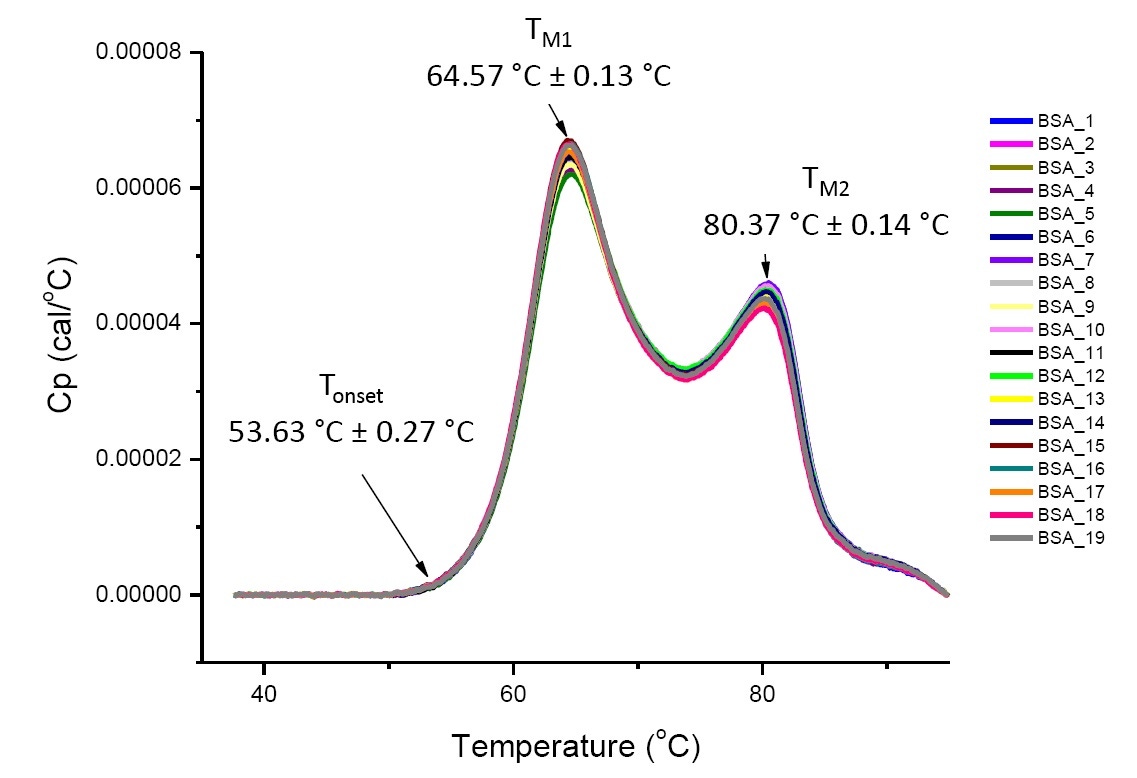
Figura 4. Dezenove termogramas de DSC de albumina sérica bovina (Sigma A1933, purificada por cromatografia) em PBS. Dados da DSC mostrados após a normalização da velocidade de varredura, subtração tampão-tampão e subtração da linha de base de integração. A média e o desvio padrão do Tinício, TM1 e TM2 são mostrados.
Dados reproduzíveis e quantitativos tornam a DSC uma ferramenta valiosa para a análise da HOS na avaliação do produto durante a fabricação (incluindo comparabilidade entre lotes e locais), na comparação de variantes de proteínas e produtos modificados (incluindo mudanças estruturais devido a glicosilação e oxidação) e na avaliação de biossimilaridade. Dados da DSC também são usados nos documentos regulatórios de apoio como uma caracterização da HOS para apresentações de novos medicamentos e biossimilares. Em uma pesquisa realizada no setor de cientistas biofarmacêuticos, a DSC foi classificada como uma ferramenta biofísica de HOS “muito útil” a “extremamente útil” para seleção de candidatos, desenvolvimento de formulação, caracterização do produto, comparabilidade e biossimilaridade[15].
Para proteínas de vários domínios como anticorpos, os termogramas DSC têm mais de uma transição de desdobramento (Figura 2). A DSC é capaz de caracterizar e quantificar os diferentes domínios e determinar os TMs para duas, três ou mais transições. Os valores de TM são obtidos a partir dos picos do termograma e podem ser simplesmente determinados a partir dos dados DSC, sem a necessidade de complexas análises de dados. Outras ensaios biofísicos que podem determinar o TM, como CD, IF e DSF, somente podem detectar o primeiro TM (na menor temperatura) ou o TM mais "dominante” paraproteínas de vários domínios. A extração de mais de um TM a partir dos dados de espectroscopia e fluorescência requer um ajuste complexo de dados e pode não ser reproduzível.
A DSC não exige mais amostras de proteínas por análise, e é possível ser menos detalhado, em comparação a outros ensaios de análise de TM. Se a amostra for limitada, uma opção seria efetuar uma classificação inicial do TM com DSF ou IF e selecionar várias amostras para validar o TM utilizando DSC. É importante validar os resultados TM com DSC e não confiar apenas na fluorescência ou espectroscopia para os ensaios de estabilidade de TM. Ensaios baseados em fluorescência podem ter artefatos que interferem com a saída e os resultados TM podem ser modificados para um valor superior (ou inferior) devido a estes artefatos. Algumas proteínas e condições de tampões não são compatíveis com fluorescência, e diferenças de TM podem não ser detectadas por esses métodos. Por fim, a fluorescência e a espectroscopia não determinam a entalpia calorimétrica e outros parâmetros termodinâmicos em comparação à DSC.
A DSC é considerada pela indústria biofarmacêutica como o estudo de estabilidade térmica “padrão-ouro” porque a DSC:
Exemplos de como dados DSC são usados na descoberta biofarmacêutica e no desenvolvimento serão encontrados neste documento, outros documentos e notas de aplicação da Malvern e nas publicações citadas.
Na fase de pesquisa de identificação de uma proteína biofarmacêutica, o foco inicial é a descoberta do destinopara possíveis interações de medicamentos. Uma vez que um alvo potencial é identificado, o primeiro passo é encontrar um medicamento que irá interagir com este alvo, que terá um efeito biológico desejado que poderia ter um bomresultado clínico de reduzir ou suprimir o estado de uma doença. Após um ou mais candidatos de medicamentos forem identificados, é importante avaliar a “capacidade de desenvolvimento" desses possíveis candidatos biofarmacêuticos.
A capacidade de desenvolvimento de um medicamento de proteína é uma avaliação das propriedades biofísicas da proteína para prever quais proteínas serão capazes de resistir às tensões que possivelmente encontrarão durante a produção de cultura de células, depuração, formulação, embalagem, envio e armazenamento de longo prazo (estabilidade). É importante entender:
A execução da análise da capacidade de desenvolvimento de um medicamento de proteínas candidato no fim de 'descoberta' antes do desenvolvimento pré-clínico começar é importante, já que, uma vez que o candidato principal for selecionado e o processo de produção for iniciado, qualquer tentativa de alterar o medicamento ou o processo terá custos crescentes e aumentará a probabilidade de o projeto ser descartado. Fazer boas escolhas nesta fase inicial resulta em menos falhas e uma redução significativa do custo para a empresa de medicamentos, bem como a produção de medicamentos de baixo custo.
Neste ponto no final descoberta/desenvolvimento, geralmente há uma quantidade limitada de proteína purificada disponível para ensaios biofísicos e um período de tempo limitado para a avaliação do ensaio de capacidade de desenvolvimento. Métodos biofísicos tais como fluorescência, difusão de luz, cromatografia de exclusão de tamanho e DSC são todos comumente usados neste ponto. Outros ensaios analíticos como ultracentrifugação e troca de hidrogênio-deutério com espectrometria de massa (HDX-MS) também podem fornecer informações úteis.
Início simples perfil de medicamentos de proteínas usando DSC para a classificação de valores TM podem ser muito úteis para a comparação de medicamentos candidatos para ajudar a encontrar as proteínas que têm uma melhor estabilidade, para prever estabilidade a longo prazo e evitar aquelas moléculas que têm estruturas menos termicamente estáveis. Proteínas com menores valores TM são consideradas menos desejáveis para progredir como candidatos de medicamentos, devido à maior instabilidade em função da temperatura.
Doyle et al.[16] publicou um estudo de caso usando métodos biofísicos para caracterizar candidatos Adnectin biologicamente específicos durante o processo de detecção, incluindo seleção de candidatos. Ensaios biofísicos incluídos SEC, AUC, SEC-MALS, cristalografia de raio x, SPR, DSF e DSC. A comparação de dois candidatos Adnectin por DSC mostrou que o Candidato B mostrou maior TM , e maior Tinício (ambos os indicando uma maior estabilidade térmica) em relação ao Candidato A. O Candidato B também mostrou evidências de reversibilidade de dobragem com DSC, ou seja, a amostra de proteína foi arrefecida e reaquecida na DSC, e o 2ºndciclo de aquecimento exibia a mesmo perfil DSC. Esta reversibilidade também sugere uma melhor expressão reduzida e tendência de agregar formação.
A maior classe de proteínas de biofarmacêuticos no mercado/em testes clínicos é anticorpos monoclonais. Anticorpos monoclonais normalmente mostram um termograma complexo com vários domínios em DSC (consulte a Figura 2 para obter um exemplo). O maior e mais proeminente pico de domínio é o Fab (Fragmento de encadernação de antígeno) de anticorpos. Os domínios CH 2e/ou CH 3também são normalmente encontrados. As posições relativas e TMs dos diferentes domínios dependem do anticorpo monoclonal específico, e podem variar de acordo com a subclasse e a engenharia, conforme demonstrado nos exemplos abaixo.
Em 2007, Garber e Demarest publicaram um artigo que descreve o uso de DSC para caracterizar dezessete anticorpos terapêuticos de comprimento total[17.18]. Usando MicroCal VP-Capillary DSC, os Fabs desses anticorpos exibiam transições de desdobramento térmico transições com pontos médios (TMs) variando de 57°C a 82°C. Os autores mostraram que o IgG1 foi a subclasse humana mais estável, seguido por IgG2 e IgG4, e para os exemplos usados, mostraram que as variações de estabilidade entre anticorpos e a subclasse semelhante foram derivadas do variáveis domínios. Os recursos exclusivos de cada gene V resultaram em uma variedade de estabilidades do Fab.
Alguns Fabs de anticorpos podem ser excepcionalmente estáveis, enquanto que outros podem ser relativamente instáveis. A estabilidade de Fab muito baixa do TM, como observado para vários casos incluídos na Referência 17, estava relacionada à expressão fraca/reduzida de anticorpos e maiores problemas de agregação, quando comparada aos anticorpos com maior Fab TM. Como resultado, é mais difícil purificar proteínas com um menor TM, resultando em rendimento reduzido durante o processo de desenvolvimento, bem como uma maior probabilidade de agregação durante o tempo de armazenamento. Os dados deste estudo[17] forneceram elementos que provam que os dados de estabilidade DSC são uma ferramenta útil para a seleção de candidatos para medicamentos biofarmacêuticos.
Ionescu et al.[19] gerou perfis DSC (usando MicroCal VP-Capillary DSC) de três anticorpos monoclonais IgG1 humanizados e os respectivos Fabs e fragmentos Fc (após a digestão enzimática) em pH neutro. Com algumas exceções, os termogramas mostraram que a transição com a maior entalpia experimental continha a contribuição dos fragmentos fab (semelhantes aos resultados na referência 17). Além disso, observou-se que os aparentes TMs variam significativamente, mesmo para fragmentos de fab provenientes da mesma linha germinal dos embriões humanos. Ionescu at al. propôs que se utilizem a entalpia de desdobramento como o parâmetro-chave para reconhecer o desenrolar dos acontecimentos no perfil de um anticorpo IgG1 intacto. Alguns termogramas DSC mostraram duas transições: a primeira transição que representa o Desdobramento do fragmento fab e o domínio CH2 e a segunda transição que representa o desdobramento do domínio CH3. Em outros anticorpos, a primeira transição DSC representa o domínio CH2 desdobrando, e a segunda transição representa o desdobramento do fragmento fab fragmento e o domínio CH3. Existem muitos outros casos onde o perfil DSC apresentou três transições, com o fab desdobrando em diferentes temperaturas, em comparação à possibilidade de derretimento dos domínios CH2 e CH3. Baixa estabilidade e a heterogeneidade do fragmento fab podem revelar-se problemáticos para armazenamento de longo prazo ou a coerência da produção. Os autores propuseram que compreensão total os recursos de um perfil DSC é importante para a seleção de candidatos nos primeiros estágios de desenvolvimento de recursos terapêuticos anticorpos monoclonais.
Do trabalho descrito pelo dois artigos acima, bem como outros publicados durante o mesmo período, foram formadas as bases para subsequentes estudos de caso e o uso de DSC e outros estudos da caracterização de estabilidade no âmbito da seleção de candidatos biofarmacêuticos.
Para definir mais profundamente a estabilidade térmica do medicamento candidato, as proteínas são comumente analisadas em mais de uma condição de pH para observar o surgimento de outras diferenças. Esses diferentes pHs podem representar condições encontradas na formulação e/ou em tampões de processo usados durante a fabricação do medicamento.
Jiang et al.[20] publicou um estudo de caso usando a caracterização HOS selecionada para avaliar a capacidade de desenvolvimento (possibilidade de fabrico, bem como de manter geral do produto qualidade) do dois potenciais medicamentos de anticorpos multiclonais. Anticorpos X e Y foram IgG2 mAbs em relação ao mesmo destino, que apresentou atividades biológicas semelhantes. Usando uma combinação de técnicas biofísicas (incluindo DSC, UV próximo DSC, UV CD aproximada, FTIR, DLS, espectroscopia de fluorescência e SEC), observaram:
A condição (pH 3) foi escolhido para replicar tampões usados durante a purificação de anticorpos e inativação viral, o pH 5 simulou o tampão das condições de armazenamento (formulação) e os tampões de pH 7 foram utilizados na purificação.
As análises MicroCal VP-Capillary DSC dos dois candidatos mAb em tampão de pH 7 (PBS) mostraram duas transições: domínios CH2/fab representaram a 1ª transição, enquanto que o desdobramento do domínio CH3 foi representado pela 2ª transição[20]. MAB Y foi um pouco mais termicamente estável (pH 7, com base no TMs da DSC assim como maior temperatura inicial (Tinício). No tampão pH 3 (C3N), houve apenas uma transição térmica DSC para cada candidato, sugerindo que as amostras foram já parcialmente desdobrado na temperatura inicial, devido ao pH baixo. O conjunto térmico de temperaturas de transição e entalpias de desdobramento tanto para os candidatos do pH 3, nível sensivelmente inferior aos em PBS, como esperado. No entanto, MAB X (TM de 63,2 °C) tiveram melhor estabilidade térmica de MAB Y (TM de 48,2 °C) em C3N, mesmo que MAB Y era mais termicamente estável em pH neutro. Embora este resultado atípico, é consistente com os dados UV CD aproximados, que revelaram que MAB X mantidas mais estrutura terciária a pH 3. Alterações na estabilidade térmica dos dois candidatos induzidas por pH 3 tratamento pareceram ser totalmente reversíveis seguidas pela diálise em PBS.
Em geral, os resultados DSC indicaram que ambas as proteínas eram muito estáveis a pH neutro, e a estabilidade térmica mAb Y foi superior a dos mAb X em PBS, mas mais baixa do que a do mAbX em C3N[20]. CD e DSC dados mostraram que MAB Y passaram por mais mudanças estruturais a pH 3 do que MAB X. Estabilidade térmica alterações induzidas a um pH 3, foram totalmente reversível para ambos os MAK, embora terciário mudanças estruturais parece ser pelo menos parcialmente irreversível para MAB Y. Em comparação com MAB X, MAB Y mostraram uma melhor estabilidade térmica em pH neutro e armazenamento melhorado estabilidade a 37°C na formulação buffer pelo pH 5. Em conjunto, os resultados sugerem que existem diferentes forças motrizes do processo de estabilidade (robustez a alterações no pH) e a estabilidade no armazenamento (impacto da temperatura elevada).
Porque mAb Y dobrados tiveram maior estabilidade térmica em PBS em pH 7 e melhor estabilidade em pH 5 durante o armazenamento a 37°C, mAb Y foi recomendado a ser evoluído para desenvolvimento adicional[20] . Além disso, devido à irreversibilidade de ambos as interrupções nas estruturas terciárias estrutura e, em seguida, auto-associação em PBS observada após o baixa incubação de pH , a recomendação para avançar mAb Y foi acompanhada por uma recomendação de desenvolver o processo de purificação para minimizar sua exposição de mAb Y para pH 3. Este estudo de caso demonstrou como os dados de estabilidade como parte da seleção de candidatos e desenvolvimento do processo ajuda a minimizar a quantidade de irreversível agregar e conformacionais variante produzida pelo processo, e, em última instância, aumentar o processo rendimento[20].
Mais alguns estudos de casos com DSC estabilidade térmica para avaliar e prever a capacidade de desenvolvimento estão resumidas em Satish et al.[21]
Tavakoli-Kenshe et al.[22] queria ver quais outras previsões além da estabilidade poderiam ser usados para prever a capacidade de desenvolvimento. Eles observaram cinco variantes de anticorpos IgG1 e IgG4, e usaram a rotação do dispositivo contra-facas que produziu condições diferentes em uma interface conhecido de sólido-líquido para avaliar a estabilidade nesse ambiente. Os anticorpos foram classificados por estabilidade com base no resultado do aparelho e comparados com dados de acelerada estabilidade térmica e a temperatura de fusão do domínio CH2 (TM1) do MicroCal VP-Capillary DSC para investigar complementaridade de técnica. Resultados sugerem que a técnicas são ortogonais, com métodos de dispositivo térmico com base na interação intramoleculares, e estabilidade de dispositivo, baseada no desdobramento localizado, e revelam regiões menos estáveis que potencializam a agregação. A modelagem molecular mostra os efeitos das modificações nas estruturas dos anticorpos e indicam um possível papel de conformidade Fc e Fab-Fc ao determinar a estabilidade suspensa das proteínas. Os dados do contra-facas apresentam teste como uma potencial ortogonal indicador de estabilidade, complementar à térmica tradicionais métodos DSC, permitindo que seleção de candidatos de proteínas com estabilidade melhorada para o processo de purificação.
Frequentemente na descoberta biofarmacêutica, as moléculas de proteínas de medicamentos "agitadas" ou "relacionadas" são modificadas ou criadas para melhorar suas características biofísicas e tornar a proteína mais capaz de ser desenvolvida. Após a engenharia, antes da seleção dos candidatos, é importante avaliar o efeito de diferentes proteínas projetadas para melhorar a estabilidade, para ver se a engenharia resultou em quaisquer efeitos nocivos. Um exemplo foi apresentado em uma nota de aplicação Malvern[23], onde MicroCal VP-Capillary DSC foi usado como ferramenta de triagem para anticorpos principais e dois anticorpos projetados, ajudando a prever a estabilidade térmica e a capacidade de desenvolvimento da engenharia que a constrói. Formação de agregação (via SEC) depois do armazenamento acelerado também foi estudada. A engenharia de anticorpos com menor TM correlacionada ao construir anticorpos com o montante mais elevado de agregação após a estabilidade acelerada, auxiliando a orientação da seleção de candidatos.
Demarest et al. projetou uma instabilidade de fab e usou MicroCal VP-Capillary DSC para analisar qual a melhor estabilidade das proteínas como demonstração[18, 24]. Para desenvolver fragmentos de anticorpos e estratégias de estabilização, escolheram um Fab mal-comportados que reconhece uma substância de tétano (αTT) originalmente isolada de uma fonte humana, e executaram uma campanha mutagênese para construir a estabilidade de fab. Os autores escolheram 45 resíduos de posições para randomização, e quase 4.500 colônias individuais foram atendidas e instruídas na expressão material. Sobrenadantes com Fab foram testadas em três temperaturas diferentes (70 ºC, 72 ºC e 74 ºC). Variantes que demonstraram termoestabilidade aprimorada foram testados novamente em termotolerância duplicada para confirmar suas propriedades (consulte a referência 24 para uma lista completa de mutantes de estabilização). Aproximadamente 1% das variantes na biblioteca exibiu uma termoestabilidade aprimorada. Quatorze das "ocorrências" estavam localizadas no domínio VH, e as quatro restantes no domínio VL. Esse resultado sugere que a estabilidade do Fab nativo foi limitada pela estabilidade marginal do VH. Surpreendentemente, nenhum mutante na biblioteca do domínio constante de ~2.000 membros pareceu estabilizar o Fab por inteiro. Os autores especularam que os mutantes de estabilização mutações nos constantes domínios realmente ocorriam, mas que a limitada estabilidade do domínio VH controla a temperatura na qual o fab se desenrola, e limita a capacidade para observar tais incidentes.
Foram descobertos quatro mutantes estabilizantes no domínio VL αTT [24]. A mutação do resíduo consensual VK4 W50 para Ala (o resíduo consensual de VK1) ou His foi altamente estabilizante. A histidina é raramente encontrada na posição 50 em domínios variáveis kappa humanos, sendo geralmente encontrada em domínios variáveis lambda humanos. O resíduo está próximo da interface do domínio VH/VL. Nossa teoria é de que sua contribuição para a estabilidade de Fab esteja associada ao reforço do domínio VH, já que o domínio VH em particular parece limitar a estabilidade do Fab αTT. No entanto, estudos com o isolado domínio VL sugerido mostram o contrário.
Foram gerados doze construtos contendo entre três e onze mutações estabilizantes identificadas na seleção inicial[24]. Várias combinações foram derivadas racionalmente para determinar a contribuição aparente que cada mutante fornece para a estabilidade de Fab. Introdução de várias mutações de estabilização nos Fab αTT aumentaram a expressão de fab em um experimento de transformação paralela/expressão, sugerindo que a mais estável resultou em aumento da sua expressão em células. Os melhores construtos exibiram consistentemente >aumentos de três dobras em rendimento expresso no tipo selvagem. A estabilidade de cada fab foi avaliada por DSC e dicroismo circular (CD). Os valores TM medidos foram usados para ordem de classificação de estabilidade de Fab.
Uma consideração importante foi o potencial efeito que essas mutações pode ter no funcionamento do dobramento do antígeno αTT Fab. As mutações de estabilidade térmica foram derivadas da seleção original utilizando uma ELISA quantitativa, detectando o domínio CL e o complemento da histidina no terminal C de CH1. Os autores viram que as mutações de estabilidade térmica melhoraram a aparente afinidade do αTT Fab em uma ELISA funcional ao tipo excepcional[24]. A capacidade funcional de cada variante de Fab pareceu estar diretamente correlacionada à sua estabilidade conforme descrito pelo Tm da DSC. A capacidade de correlacionar atividade funcional e expressão de proteínas para o TM do DSC sugeriu que escolher os mais estável resultará em uma melhoria na capacidade de desenvolvimento.
Seeliger et al.[25] incorporou estratégias de design computacional para sistematicamente modificar um anticorpo que exibia uma tendência para agregar in vitro. A série resultante de anticorpos estreitamente relacionados concebidos a partir um tipo excepcional (WT) mAb1 mostraram uma melhor estabilidade conforme avaliada por métodos biofísicos (incluindo MicroCal VP-Capillary DSC) bem como experimentos de estabilidade a longo prazo. Conforme observado por Demarest et al. observou na exemplo acima com αTT Fab[24], que níveis de expressão mutantes de proteína também melhoraram em comparação ao candidato de tipo excepcional. Em conjunto, os dados experimentais e computacionais utilizados para a realização deste estudo demonstram como métodos computacionais podem ser usados para guiar a otimização de anticorpos para estabilidade.
DSC da proteína original tipo excepcional apresentava um TMde 68 °C para a primeira transição do termograma (identificado como o domínio fab). Em termogramas DSC para mutações de cadeia pesada ou cadeia leve, a TM para a primeira transição térmica (fab) DSC aumentou para 68,9°C - 72,8°C. No mutante projetado para cadeiras pesadas e leves, a primeira endoderme é observada a 70,5°C, com o maior pico (fab) a 83,5°C. O TMpara o desdobramento dos anticorpos Fab aumenta cerca de 16 K após a remoção das 'responsabilidades' previstas na cadeia pesada e na cadeia leve. Durante a manipulação foi monitorado via a alteração na fluorescência triptofana, a mesma tendência foi observada[ 25] ..
A simulação em computador previu que a maioria das mutações introduzidas aumentam a estabilidade termodinâmica. Esses resultados estavam em linha com os dados obtidos junto dos CDS e em ângulo reto difusão de luz (RAL). Ambos os experimentos demonstraram um aumento contínuo de estabilidade com o aumento do número de mutações. De ambos os experimentos, os autores também descobriram que a engenharia da cadeia leve tinha um nítido efeito sobre a estabilidade, o que está de acordo com os calculado as alterações nas termoestabilidade para cada mutação[25].
Tidas as variantes projetadas do mAb1 foram expressadas com títulos acima de 50 mg/L, ao passo que com tipo excepcional, os títulos foram abaixo de 1 mg/L. O dramático aumento da expressão de níveis para as variantes com apenas uma corrente de engenharia não foram previsíveis dos resultados DSC, que mostraram um pequeno aumento na estabilidade durante essas variantes. No entanto, os cálculos sugerem que a estabilidade dos domínios de imunoglobulina individuais é aprimorada com as mutações introduzidas. Em experimentos que envolvam aquecimento, é plausível que a estabilidade de cada cadeia é o fator limitante, indicando que os efeitos observados foram cinética em vez de equilíbrio termodinâmico, próprio efeitos. Assim que um domínio se desenrola, o efeito estabilizador da complexa formação desaparece, e, como consequência, a segunda cadeia começa a desenrolar-se também. De produção de proteínas e dobragem in vivo, a presença de um domínio dobrado de forma estável para servir como um nucleação para uma corrente menos estável poderá ser suficiente para aumentar substancialmente a quantidade de anticorpos dobrados com êxito[25] .
Na perspectiva de uma empresa que desenvolve um medicamento biofarmacêutico medicamento, o passo-a-passo para a maior estabilidade de proteínas (do DSC e outros ensaios biofísicos), traduzir em passo-a-passo melhora a longo prazo a estabilidade e a melhoria de vida útil? Os autores realizaram um acelerado estudo de estabilidade a 40°C, contendo as variantes projetadas, e os autores observaram melhorias substanciais de estabilidade a longo prazo, tal como avaliada pelo conteúdo de SEC. A variante projetada foi também a mais estável por todo o período de 4 meses de testes[23]. A estabilidade termodinâmica dos domínios variáveis certamente é um fator importante para a estabilidade a longo prazo. Mas também é evidente nos dados dos autores, assim como em outros dados publicados, que a estabilidade termodinâmica e de vida útil não são fáceis de correlacionar em cada caso, sendo, podem ter ajustes feitos para melhorar a estabilidade na fase da formulação[25,26,27,28].
Publicações recentes descrevem a engenharia racional e o uso de DSS e outras ferramentas para caracterizar a estabilidade das proteínas, incluindo:
O material de referência de anticorpos multiclonais do National Institute of Standards and Technology (NISTmAb)[35] foi avaliado em capacidade de desenvolvimento com ferramentas biofísicas HOS geralmente usadas para medicamentos mAb candidatos. O NISTmAb não se destina a ser um terapêutica molécula, e a avaliação da capacidade de desenvolvimento do NISTmAb foi tirada como particulares para as diversas classes, representante IgG1 MAB[36]. O NISTmAb foi avaliado em diferentes condições de tampão de estabilidade e integridade eletroforese, e métodos SEC-MALS, DLS e DSC. A estabilidade térmica do NISTmAb amostra no tampão PBS era determinado pelo MicroCal VP-Capillary DSC. O exemplo NIST mostra três principais transições térmicas atribuídas como o CH 2 por 71,2 °C, o CH3 por 84,1 °C, e o domínio fab a 88,9 °C.
Em comparação com os perfis DSC de moléculas IgG1 da referência 17, o NIST Fab é mais estável (88,9 °C); portanto, concluiu-se que a molécula tem um favorável perfil de HOS[36]. Com base na completa análise biofísico do conjunto de dados, o NISTmAb tem uma capacidade de desenvolvimento favorável e as mudanças observadas na base molecular após condições de estresse não representam os riscos que não podem ser corrigidos com a direita formulação e fabricação na última fase do processo de desenvolvimento.
Resultados apresentados neste documento técnico demonstram claramente a importância e a eficácia de incorporação DSC como um biofísico experimento estabilidade durante a biofarmacêutica seleção dos candidatos. Usando os resultados de DSC, juntamente com outros estudos de estabilidade, as empresas de produtos biofarmacêuticos podem tomar decisões informadas sobre as formulações mais estáveis e com maior capacidade de desenvolvimento, ou seja, aquelas com melhores chances de produção e purificação; e aquelas com menos probabilidade de apresentar problemas de estabilidade a longo prazo e agregação em sua formulação final e como um medicamento. Isso resulta em uma produção de medicamentos mais econômica e uma maior probabilidade de que a formulação final do medicamento permaneça ativa, estável, segura e na conformação de dobramento correta.
Analytical Techniques for Biopharmaceutical Development, R. Rodriguez-Diaz, T. Wehr, S. Tuck (eds.), Taylor & Francis, New York USA (2005).
Biophysical Characterization of Proteins in Developing Biopharmaceuticals, D.J. Houde, S.A. Berkowitz (eds.), Elsevier, Amsterdam, Netherlands (2015).
Biophysical Methods for Biotherapeutics: Discovery and Development Applications, T.K. Das (ed.) John Wiley & Sons, Hoboken NJ USA (2014).
Biophysics for Therapeutic Protein Development, L.O. Nahri (ed.), Springer New York, USA (2013).
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 1. Monoclonal Antibody Therapeutics: Structure, Function, and Regulatory Space, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1176 (2014). doi: 10.1021/bk-2014-1176.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 2. Biopharmaceutical Characterization: The NISTmAb Case Study, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1201 (2015). doi: 10.1021/bk-2015-1201.
State-of-the-Art and Emerging Technologies for Therapeutic Monoclonal Antibody Characterization Volume 3. Defining the Next Generation of Analytical and Biophysical Techniques, J.E. Schiel, D.D. Davis, O.V. Borisov (eds.), ACS Symposium Series Vol 1202 (2015) DOI: 10.1021/bk-2015-1202.